TI/Matplotlib + numpy
Krótkie wprowadzenie do biblioteki NumPy pojawiło się już w zeszłym roku.
Spis treści
Wycinki
Fragment z
http://en.wikibooks.org/wiki/Non-Programmer%27s_Tutorial_for_Python_2.0/Revenge_of_the_Strings, autorstwa wikibooksen:User:Jrincayc, wikibooksen:User:Whiteknight, wikibooksen:User:Siebengang, wikibooksen:User:33rogers.
Z poprawkami!
Indeksy
Indeksując napis odwołujemy się do wybranego znaku. Znaki mogą być liczone od początku napisu (pierwszy ma numer 0) lub od jego końca (ostatni ma numer –1).
| 0 | 1 | 2 | ... | –2 | –1 | |||||||||
| ↓ | ↓ | ↓ | ↓ | ↓ | ↓ | |||||||||
| tekst = | " | S | T | R | I | N | G | " | ||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ↑ | ↑ | |||||||||||||
| [ | ] |
Na powyższym rysunku czerwone indeksy są liczone od początku napisu, a niebieskie od jego końca.
Teraz przykłady:
| tekst[0] | → | "S" |
| tekst[1] | → | "T" |
| tekst[2] | → | "R" |
| tekst[−2] | → | "N" |
| tekst[−0] | → | "S" |
| tekst[20] | → | red|IndexError |
Nie ma indeksu −0, bo w Pythonie −0 == 0, więc −0 również wskazuje na początek napisu.
Numery wskazujące na miejsca poza końcem napisu lub przed jego początkiem są błędne, Python zwróci informację o błędzie, tzw. IndexError.
Przykład — indeksy dla napisu "string"
Indeksy dodatnie
>>> tekst[0]
's'
>>> tekst[1]
't'
>>> tekst[2]
'r'
>>> tekst[3]
'i'
>>> tekst[4]
'n'
>>> tekst[5]
'g'
Indeksy ujemne
>>> tekst = "string"
>>> tekst[-1]
'g'
>>> tekst[-2]
'n'
>>> tekst[-3]
'i'
>>> tekst[-5]
't'
>>> tekst[-4]
'r'
>>> tekst[-5]
't'
>>> tekst[-6]
's'
Wycinki
Pobranie wycinka sekwencji polega na wybraniu jej fragmentu — podsekwencji. Miejsca początku i końca zakresu z którego pobieramy wycinek liczone są podobnie jak przypadku indeksowania. Istotna różnica jest taka, że wskazujemy podsekwencję, a nie pojedynczy element — i to w sposób szczególny. Przedział który wskazujemy jako [a:b], jest zamknięty od strony a, a otwarty od strony b.
Lewy i prawy kraniec wycinka podajemy oddzielając indeksy dwukropkiem :. Każdą z tych wartości można pominąć, w skrajnym przypadku zostawiając tylko dwukropek. Jeśli dany indeks pominiemy, zostaną użyte wartości domyślne:
- domyślne położenie początku wycinka to początek sekwencji
- domyślne położenie końca wycinka to koniec całej sekwencji
Taka konwencja ma pewne zalety, w szczególności dla dowolnego indeksu a prawdą jest, że
sekwencja[:a] + sekwencja[a:] == sekwencja
Konstruując wycinek możemy pobierać co n-ty element napisu. Robi się to podając wartość kroku n po kolejnym dwukropku. Domyślnie n ma wartość 1.
Teraz przykłady:
| tekst[1:4] | → | "TRI" |
| tekst[:5] | → | "STRIN" |
| tekst[:–1] | → | "STRIN" |
| tekst[–4:] | → | "RING" |
| tekst[:] | → | "STRING" (tzw. pełny wycinek, czyli kopia sekwencji) |
| tekst[::–1] | → | "GNIRTS" |
| tekst[−10:−2] | → | "STRI" |
Numery wskazujące na miejsca poza rozmiarem napisu nie są błędne, Python wybierze tylko istniejące znaki.
Numpy
Tablice
W zeszłym roku była mowa o tablicach (arrays) w numpy. Ograniczaliśmy się praktycznie do przypadku dwuwymiarowego, czyli macierzy. Warto sobie uświadomić, że obiekt array może być wielowymiarową tablicą. Przytoczymy teraz kilka przydatnych funkcji dotyczących tablic w numpy.
import numpy as np
a = np.ones((2,3,4)) # tworzy tablicę jedynek o wymiarze 2x3x4
a = np.empty((2,3)) # tworzy pustą tablicę o wymiarze 2x3
a = np.zeros((2,3,4,5)) # twrzy tablicę zer o wymiarze 2x3x4x5
# tak, w numpy można tworzyć więcej niż 3-wymiarowe tablice!
# jeśli potrzebowalibyśmy bardzo dużo wymiarów, a nie chce nam się ich po kolei wpisywać możemy zrobić tak:
a = np.empty(tuple([10 for i in range(20)])) # 20-sto wymiarowa tablica, w której każdy wymiar ma długość 10
#np.empty przyjmuje jako argument obiekt tuple, dlatego utworzyliśmy w wygodny sposób listę wymiarów, a następnie przerabiamy ją na tuple, żeby podać jako argument
# łatwiejszym do wyobrażenia, 3-wymiarowym odpowiednikiem tego jest:
a = np.empty(tuple([10 for i in range(3)])) # 3-wymiarowa tablica, w której każdy z wymiarów ma długość 10
A = np.random.randint(10,size = (3,3)) # wytwarza tablicę losowych elementów całkowitych o wymiarach 3x3 (takie tablicę częst się przydają jako dane testowe do naszych funkcji/programów)
A = np.random.randint(10,size = (3,3,5)) # wytwarza tablicę analogiczną do powyższej, ale o wymiarze 3x3x5
wiersze,kolumny = numpy.shape(A) # co się stanie, gdy A ma wymiar mniejszy/większy niż 2?
- Jaki będzie wynik następujących instrukcji:
a = np.arange(1,10)
a = np.arange(10)
a = np.arange(1., 10)
a = np.arange(1, 10, 2.5)
a = np.arange(1,2,10)
- Utwórz 3-wymiarową tablicę A, o wymiarach 10x5x3 taką, że A[i,j,k] = "i,j,k" (wartością każdego elementu będzie string zawierający wszystkie indeksy). Aby zainicjalizować poprawnie tablicę pamiętaj o wybraniu odpowiedniego typu zmiennych (dtype). Wypisz ją, przyjrzyj się w jakiej kolejności są wypisywane elementy, gdy napiszesz po prostu.
print A
- Utwórz 100-wymiarową tablicę zer, o każdym wymiarze = 10. Ile maksymalnie wymiarów może mieć tablica w numpy?
- Z listy [[1, 2, 3], [4, 5, 6],[7, 8, 9]] utwórz macierz.
- Zastanów się, jak zmienić swoją funkcję obliczającą splot, tak, żeby radziła sobie też z wektorami różnej długości.
Trochę wiadomości o tablicach w numpy
- W numpy jak i w Pythonie obowiązuje numeracja od zera — stąd pierwszy element wektora a o długości N to a[0], a ostatni a[N-1].
- Elementy tablicy indeksuje się: [math]A[i_0,i_1,...,i_{n-1}][/math], gdzie [math]i_j[/math] to indeks w wymiarze j. Tzn dla tablicy dwuwymiarowej: A[wiersz, kolumna].
- Łączenie tablic wykonuje się za pomocą funkcji numpy.concatenate((a1, a2, ...), axis=0). a1, a2, ... oznacza tablice, a axis oś, w której tablice zostaną połączone. Parametr ten ma domyślnie wartość 0. Tablice muszą mieć taki sam rozmiar w osi, w której będą połączone. Funkcja zwraca tablicę wynikową.
- Usuwanie elementu z tablicy (wielowymiarowej):
dane2 = dane[numpy.r_[:i,i+1:dane_1.shape[0]]]
Problemy
Proste operacje
from numpy import *
l1 = [1,1,1]
12 = ones(3)
Jaki będzie wynik następujących operacji:
l1 * 3
l2 * 3
Jakie operacje należy wykonać na liście l1, aby otrzymać wynik analogiczny do l2*3?
l1 / 3
l2 / 3
Plansza kółko-krzyżyk
Zaprojektuj tablicę złużącą do gry w kółko i krzyżyk w trzech wymiarach. Kółka to zera, krzyżyki to jedynki. To znaczy zdefiniuj kilka funkcji, które będą umożliwiały grę w kółko i krzyżyk w trzech wymiarach:
- funkcję tworzącą pustą planszę
- funkcję stawiąjącą krzyżyk w wybranym miejscu(określonym współrzędnymi x,y,z)
- funkcję stawiąjącą kółko w wybranym miejscu
- funkcję wypisującą aktualny stan planszy
- Rozszerz poprzednie zadanie o wyrysowywanie trójwymiarowe stanu planszy.
- Utwórz ze zdefiniowanych funkcji moduł
Dopasowanie metodą najmniejsztch kwadratów
W pracy doświadczalnej wielokrotnie badamy zależność z jaką wielkość [math]y[/math] zależy od wielkości [math]x[/math]. Zwykle ta zależność fizyczna jest postaci:
gdzie [math]f[/math] — zależność fizyczna, [math]par[/math] — parametry.
Żeby zobaczyć, czy funkcja [math]f(x;par)[/math] dobrze oddaje realną zależność fizyczną między [math]y[/math] a [math]x[/math] obliczamy suma kwadratów residuów ([math]R[/math]) — sumę kwadratów różnic wyników pomiaru i ich wartości oczekiwanych, wyliczonych z (Equation 1):
gdzie para [math](x_i,y_i)[/math] oznacza wynik piątego pomiaru wielkości [math]x[/math] i [math]y[/math].
Szukając parametrów [math]par[/math] (Equation 1), wybieramy te, dla których [math]R[/math] jest najmniejsze. Jest wiele różnych algorytmów, którymi można się w tym posłużyć. Można też po prostu przeskanować przestrzeń parametrów, w szczególności, kiedy wiemy, jakie są fizyczne zakresy wartości tychże; i wybrać takie, które minimalizują [math]R[/math].
Błąd dopasowania wybranego już parametru [math]\theta[/math] przedstawia się następującym wzorem:
Zadanie
Napisz moduł zawierający funkcję do obliczenia sumy kwadratów residuów (Equation 2) i błędu dopasowania parameterów (Equation 3).
- Załóż, że funcja opisująca relację między danymi ma postać (Equation 1) .
func(dane,parametry)
- Dane znajdują się w macierzy o wymiarach n×2 (dane).
- Funkcja obliczająca (Equation 2) powinna mieć przekazywane dane w wektorze o wymiarach (dane), nazwę funkcji charakteryzującej zależność fizyczną i służącej do obliczania wartości oczekiwanych (func) i listę parametrów funkcji (params):Pamietaj o rozpakowaniu listy z parametrami funkcji func.
def chi2(dane, func, params):
- Funkcja obliczająca (Equation 3) powinna zwracać niepewności wszystkich podanych w liście parametrów. Parametrem opcjonalnym powinno być [math]\Delta[/math], służące do obliczenia drugiej pochodnej (patrz wskazówka 1). Funkcja powinna korzystać z funkcji chi2. Nagłówek funkcji:
def delta_p(dane, func, params,delta=0.000001):
- Metodą brute force, skanując przestrzeń parametrów, sprawdź, czy do danych pasuje zależność (Equation 4) (opis danych i parametrów w tytule rysunku).
- Zobacz na wykresie, jak dobre jest Twoje dopasowanie.
- Zmodyfikuj funkcję, tak żeby mogła uwzględnić niepewności pomiarowe w obliczaniu sumy kwadratów residuów: [math]R = \sum_{i=1}^N \left( \frac{y_i-f(x_i;par)}{\sigma_i}\right)^2[/math], gdzie [math]\sigma_i[/math] i-tą niepewność pomiarową [math]\mathrm{pomiar}_i[/math]. Załóż, że jeżeli tablica z danymi ma trzy kolumny, w trzeciej znajdują się niepewności pomiarowe [math]\delta y[/math]. Jeżeli tablica z danymi ma cztery kolumny, w czwartej znajdują się niepewności pomiarowe y-ka — [math]\delta y[/math], a w trzeciej niepewności pomiarowe x-a — [math]\delta x[/math].
Dane znajdują się w Plik:Dane.txt. Pierwsza kolumna przedstawia interwały [math]T[/math]. Druga obliczone wartości wzmocnienia synaptycznego FR, które może być opisywane następującą zależnością:
- [math]\tau_\mathrm{f} = \unit{5 - 100}{ms}[/math]
- [math]U_\mathrm{SE} = 0.001 - 0.03[/math]
W jakim zakresie Delta wynik niepewności jest stabilny?
Wskazówka 1
Numeryczne obliczanie pierwszej pochodnej:
- [math]\frac{\mathrm d x}{\mathrm dt} = \frac{x(t+\Delta t)-x(t)}{\Delta t}[/math]
Numeryczne obliczanie drugiej pochodnej:
- [math]\frac{\mathrm d^2 x}{\mathrm dt^2} = \frac{f'(x)-f'(x-\Delta t)}{\Delta t}=\frac{x(t+\Delta t) +x(t-\Delta t) -2x(t)}{\Delta t^2}[/math]
Wskazówka 2
Skorzystaj z modułu Numpy do wczytania danych (loadtxt) oraz obliczenia wartości funkcji wykładniczej (exp).
Zadanie_z_chi2/rozwiązanie macierzowe
Najpierw definiujemy funkcję służącą do obliczania FR na podstawie modelu teoretycznego:
from __future__ import division
import numpy
def FRobl(tau, Use, T):
return 1 + (1 + (1 - Use)*numpy.exp(-T/tau))**4
Proszę zwrócić uwagę, że tak zdefiniowana funkcja działa równie dobrze dla zwykłych liczb, jak i dla tablic numpy.ndarray, o ile podane argumenty obsługują działania wykorzystywane w funkcji, czyli /,
*, **, -, + i numpy.exp.
Z kolei __future__.division jest importowane bo chce się upewnić, że dzielenie zawsze jest zwykłym dzieleniem, nawet jeśli funkcja zostanie wywołana jako FRobl(2, 0.03, 1).
Następnie definiujemy funkcje do obliczania pierwszej i drugiej pochodnej:
def pochodna(f, i, *params, **opts):
eps = opts.get('eps', 1e-6)
params_eps = list(params)
params_eps[i] += eps
return (f(*params_eps) - f(*params))/eps
def pochodna2(f, i, *params, **opts):
eps = opts.get('eps', 1e-6)
params_eps = list(params)
params_eps[i] += eps
return (pochodna(f, i, *params_eps) -
pochodna(f, i, *params)) / eps
Jeśli się pamięta wzór na pochodną, to tutaj nie ma wielkich niespodzianek.
Jak działa *params, **opts w liście parametrów?
Mamy argument params, który zawierać będzie wszystkie nadmiarowe argumenty pozycyjne, oraz opts który zawierać będzie wszystkie nazwane argumenty. Dzięki temu możemy funkcję wywołać z dowolną liczbą parametrów params, które zostaną przekazane do f.
Do opts trafiają argumenty nazwane. Jeśli napiszemy eps=..., to nasz argument zostanie wydobyty ze słownika i użyty jako eps. Jeśli nie podamy argumentu eps=..., to jako eps zostanie użyty drugi argument do get, czyli 1e-6. Jeśli podamy argumenty nazwane inne niż eps=... to zostaną one zignorowane -- niestety nasza funkcja nie jest doskonała.
Aby obliczyć niepewność parametru, musimy mieć funkcję obliczającą rms (rmsfunc) i miejsce w którym liczymy (params) i numer parametru który nas interesuje (i).
def niepewnosc(rmsfunc, i, *params):
return (pochodna2(rmsfunc, i, *params)/2)**-0.5
W naszym wypadku funkcja obliczająca rms może wyglądać tak:
def frms(T, FR, tau, Use):
FR2 = FRobl(tau, Use, T)
return ((FR2 - FR)**2).mean()**0.5
Sens jest taki, że obliczamy co daje model teoretyczny FRobl jak dla danego zestawu ([math]\tau[/math], [math]U_{SE}[/math]) = (tau, Use) i pewnego T i porównujemy z wynikami doświadczalnymi FR. Otrzymujemy zestaw odchyleń, a rms jest oczywiście pierwiastkiem ze średniej kwadratów tych odchyleń.
Przechodąc do meritum, możemy albo się posłużyć danymi doświadczalnymi, albo np. wygenerować dane dla znanych ($\tau$, $U_{SE}$). To drugie podejście pozwala nam sprawdzić naszą metodę, bo wiemy dokładnie jaki wynik powinniśmy otrzymać.
Zestaw danych wygenerowanych wraz z parametrami:
def dane_symulowane(tau, Use):
T = numpy.arange(5, 80, 5)
return T, FRobl(tau, Use, T), tau, Use
T_opt, FR_opt, tau_opt, Use_opt = dane_symulowane(30, 0.02)
Dopasowanie wykonujemy metodą brute force, czyli po prostu przeszukujemy całą przestrzeń parametrów. Implementację rozbijmy na dwie części — najpierw obliczanie rms, potem znajdywanie minimum:
def rms_brute_force(T, FR):
tau = numpy.arange(5, 101, 1)
Use = numpy.arange(0.001, 0.031, 0.001)
# tau3 = tau
# Use3 = Use[:, numpy.newaxis]
# T3 = T[:, numpy.newaxis, numpy.newaxis]
tau3 = tau[numpy.newaxis, numpy.newaxis, :]
Use3 = Use[numpy.newaxis, :, numpy.newaxis]
T3 = T[:, numpy.newaxis, numpy.newaxis]
print('kształty tau, Use, T: ',
tau3.shape, Use3.shape, T3.shape)
wynik = FRobl(tau3, Use3, T3)
FR3 = FR[:, numpy.newaxis, numpy.newaxis]
odchylenie = wynik - FR3
rms = (odchylenie**2).mean(axis=0)
return tau, Use, rms
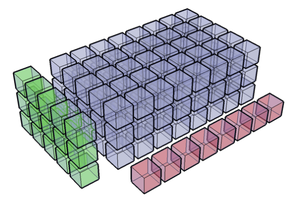
Do szybkiego przeprowadzenia obliczeń na całej siatce wykorzystujemy broadcasting. Tworzymy trzy wektory, czyli tablice o długości większej niż 1 w jednym tylko kierunku — (1, 1, M2), (1, M1, 1), (N, 1, 1). Każdy z wektorów jest skierowany w inną stronę, więc po wykonaniu działania element po elemencie (tak jak to się dzieje w FRobl), otrzymujemy macierz o wymiarze (N, M1, M2).
def dopasowanie_brute_force(T, FR):
tau, Use, rms = rms_brute_force(T, FR)
ind_min = numpy.unravel_index(rms.argmin(), rms.shape)
Use_min = Use[ind_min[0]]
tau_min = tau[ind_min[1]]
print('minimum dopasowania to τ={} Use={}'.format(tau_min, Use_min))
assert frms(T, FR, tau_min, Use_min) == rms.min()
D_tau_min = niepewnosc(frms, 2, *(T, FR, tau_min, Use_min))
D_Use_min = niepewnosc(frms, 3, *(T, FR, tau_min, Use_min))
print('niepewności wynoszą Δτ={} ΔUse={}'.format(D_Use_min, D_tau_min))
return tau, Use, rms, tau_min, D_tau_min, Use_min, D_Use_min
Aby znaleźć optymalne wartości parametrów, czyli takie dla których rms jest najmniejszy, wykorzystujemy numpy.argmin. Ta funkcja nam zwraca indeks minimalnego elementu, ale po "spłaszczeniu". Numer wiersza i kolumny uzyskujmy przez numpy.unravel_index.
Do obliczenia niepewności mamy zdefiniowaną wcześniej funkcję niepewnosc
Wynik:
tau, Use, rms, tau_min, D_tau_min, Use_min, D_use_min = dopasowanie_brute_force(T_opt, FR_opt)
</tt>
Wyniki możemy narysować przy pomocy następujących funkcji, które zostawie prawie bez komentarza:
<source lang=python>
from __future__ import unicode_literals
from matplotlib import pyplot, patches
__name__ == '__main__' and pyplot.ion()
# unicode_literals powoduje, że matplotlib wie, że wszystkie stringi to Unicode
# pyplot.ion() podowuje coś
def plot_map(tau, Use, rms):
f = pyplot.figure()
ax = f.add_subplot(111)
X, Y = numpy.meshgrid(tau, Use)
map = ax.pcolor(X, Y, rms)
ax.set_xlabel(r'tau')
ax.set_ylabel(r'Use')
f.colorbar(map)
f.show()
return f
def plot_map2(tau, Use, rms, **kwargs):
f = pyplot.figure()
ax = f.add_subplot(111)
X, Y = numpy.meshgrid(tau, Use)
cont = ax.contour(X, Y, rms)
ax.set_xlabel(r'tau')
ax.set_ylabel(r'Use')
f.colorbar(cont)
ax.clabel(cont, inline=1, **kwargs)
f.show()
return f
def plot_dane(T, FR, label1, T2, FR2, label2):
f = pyplot.figure()
ax = f.add_subplot(111)
ax.set_xlabel(r'T')
ax.set_ylabel(r'FR')
ax.plot(T, FR, 'ro', label=label1)
ax.plot(T2, FR2, '-g', label=label2)
ax.legend(loc='best')
return f
def plot_dopasowanie(T, FR, tau, Use):
T2 = numpy.linspace(T.min() - 1, T.max() + 10, 300)
FR2 = FRobl(tau, Use, T2)
return plot_dane(T, FR, 'dane', T2, FR2, 'dopasowanie')
I na koniec rysunek z zeznaczonym rms:
def plot_dopasowanie_map(tau, Use, rms,
tau_min, D_tau_min, Use_min, D_Use_min):
f = plot_map(tau, Use, rms)
ax = f.gca()
ax.plot(tau_min, Use_min, 'xy', markersize=5)
ellip = patches.Ellipse(xy=(tau_min, Use_min),
width=D_tau_min, height=D_Use_min)
ellip.set_facecolor('none')
ellip.set_linestyle('dashed')
ellip.set_edgecolor('yellow')
ax.add_artist(ellip)
return f
Działania macierzowe
Broadcasting (dopełnianie) jest słowem, które określa, jak numpy traktuje wektory o różnym kształcie. Przy spełnieniu pewnych warunków, mniejszy wektor jest "rozszerzony" (powiększony) tak, żeby mieć kształt korespondujący z kształtem większego wektora — odpowiednie wymiary zostają dopełnione. Dopełnienie umożliwia przeprowadzenie operacji macierzowych tak, że pętle są wykonywane w C, a nie w Pythonie, co zwykle prowadzi do bardziej efektywnych implementacji algorytmów.
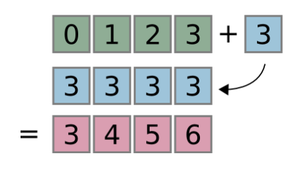
Najprostszym przypadkiem broadcastingu jest dodawanie liczby (skalara) do macierzy. W tej sytuacji, każdy element macierzy zostanie zwiększony o tęże liczbę. Przyjrzyj się następującemu przykładowi i obrazkowi Figure 1:
>>> x = np.arange(4)
>>> x = array([0, 1, 2, 3])
>>> x + 3
array([3, 4, 5, 6])
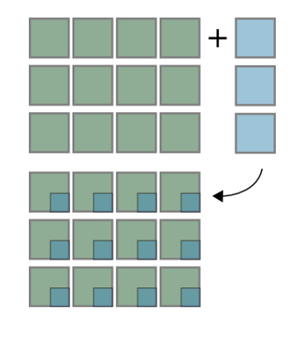
W przypadku więcejwymiarowym, oś, która powinna zostać dodana — w którą stronę wektor powinien zostać dopełniony, zostaje zaznaczona explicite słowem np.newaxis (albo None). Tak, jak na poniższym przykładzie i Rys. Figure 2:
>>> a = np.arange(12).reshape((3,4))
>>> b = np.array([1,2,3])[:,np.newaxis]
>>> a + b
array([[ 1, 2, 3, 4],
[ 6, 7, 8, 9],
[11, 12, 13, 14]])
Przypadek trójwymiarowy (Rys. Figure 3):
>>> x = np.zeros((3, 5))
>>> y = np.zeros(8)
>>> (x[..., None] + y).shape
(3, 5, 8)
Można tez skorzystać z funkcji np.broadcast_arrays.
Zadanie 1
Przed przystąpieniem do zadania polecamy zapoznać się z przykładami stąd
Zadanie 2
Przepisz operacje zachodzące w pętlach i funkcjach w zadaniu z dopasowaniem metodą najmniejszych kwadratów tak, żeby skorzystać z operacji macierzowych.
Żeby znaleźć najmniejszą wartość w macierzy z kwadratami residuów skorzystaj z metody min() strunktury ndarray. Żeby uzyskać spłaszczony indeks tego elementu skorzystaj z metody argmin() strunktury ndarray i następnie z funkcji numpy.unravel_index(indices, dims, order='C'), żeby znaleźć wartości parametrów, które minimalizują sumę kwadratów residuów.
Deklarowanie własnych typów danych
Deklarowanie własnych typów danych odbywa się za pomocą funkcji dtype.
Przykładowo (przykłady z dokumentacji):
>>> np.dtype([('f1', np.int16)])
dtype([('f1', '<i2')])
Daje nam rekord o polu 'f1' o typie danych int16.
np.dtype([('f1', np.uint), ('f2', np.int32)])
dtype([('f1', '<u4'), ('f2', '<i4')])
Daje nam dwa pola rekordu jedno typu unsigned int, drugie typu int32.
Można też zadeklarować rekord zawierający liczbę typu double i string dziesięciobajtowy:
np.dtype([('a','f8'),('b','S10')])
dtype([('a', '<f8'), ('b', '|S10')])
Przykład użycia:
>>> typ = np.dtype([("a", np.int16), ("b", "|S10")])
>>> typ
dtype([('a', '<i2'), ('b', '|S10')])
>>> a = np.array([(5, "napis")], dtype = typ)
>>> a
array([(5, 'napis')],
dtype=[('a', '<i2'), ('b', '|S10')])
>>> a = np.array([("n", "napis")], dtype = typ)
Traceback (most recent call last):
File "<pyshell#171>", line 1, in <module>
a = np.array([("n", "napis")], dtype = typ)
TypeError: expected a readable buffer object
>>> a = np.array([(5, 5)], dtype = typ)
>>> a
array([(5, '5')],
dtype=[('a', '<i2'), ('b', '|S10')])
>>> a = np.array([(5,)], dtype = typ)
Traceback (most recent call last):
File "<pyshell#174>", line 1, in <module>
a = np.array([(5,)], dtype = typ)
TypeError: expected a readable buffer object
>>> typ2
dtype([('c', '<i2')])
>>> typ2 = np.dtype([("c", (np.int16, 3))])
>>> a = np.array([((3.5,1),)], dtype = typ2)
Traceback (most recent call last):
File "<pyshell#218>", line 1, in <module>
a = np.array([((3.5,1),)], dtype = typ2)
TypeError: expected a readable buffer object
>>> a = np.array([((3,3,3),)], dtype = typ2)
>>> a
array([([3, 3, 3],)],
dtype=[('c', '<i2', 3)])
>>> typ3 = np.dtype([("c", (np.int16, 5))])
>>> a = np.array([((3,3,4,5),)], dtype = typ3)
Traceback (most recent call last):
File "<pyshell#225>", line 1, in <module>
a = np.array([((3,3,4,5),)], dtype = typ3)
TypeError: expected a readable buffer object
>>> a = np.array([((3,3,4,5,6),)], dtype = typ3)
>>> a
array([([3, 3, 4, 5, 6],)],
dtype=[('c', '<i2', 5)])
Zadanie 1
Pod linkiem znajduje się plik tekstowy zawierający dane o triggerach z jednego z eksperymentów. Obejrzyj dane. Zaprojektuj za pomocą funkcji dtype z modułu numpy typ danych, który opisuję dane, znajdujące się w rzędach. Następnie wczytaj je za pomocą funkcji loadtxt, pamietając o opuszczeniu pierwszej linii i zadaniu przecinków, jako separatorów między kolumnami i wypisz wektor z danymi. Program nie powinien mieć więcej niż 5 linii.
Zadanie 2
Wczytaj dane z poprzedniego zadania i spod łącza, a następnie wypisz średnie czasy odpowiedzi dla bodźców typu pozytywnego, neutralnego i negatywnego