"Programowanie dla Fizyków Medycznych"/DICOM: Różnice pomiędzy wersjami
| Linia 313: | Linia 313: | ||
Główny program powinien wyświetlać obraz, utworzyć dla niego obiekt do operacji i wywoływać odpowiednio jego metody zależnie od klawiszy naciśniętych przez użytkownika. | Główny program powinien wyświetlać obraz, utworzyć dla niego obiekt do operacji i wywoływać odpowiednio jego metody zależnie od klawiszy naciśniętych przez użytkownika. | ||
| − | + | <source lang='python'> | |
| − | + | # -*- coding: utf-8 -*- | |
| − | + | """ | |
| − | + | Created on Wed Jan 10 16:09:55 2018 | |
| + | |||
| + | @author: user | ||
| + | """ | ||
| + | |||
| + | import pydicom as dcm | ||
| + | import numpy as np | ||
| + | import cv2 | ||
| + | |||
| + | filename="C:/Users/user/Documents/z_programowanie/DICOM/LADJAN_MRI/localizer - 1/IM-0001-0001.dcm" | ||
| + | dataset = dcm.dicomio.read_file(filename) | ||
| + | |||
| + | def nothing (x): | ||
| + | pass | ||
| + | |||
| + | def lin(arr, a, b, maks=255): | ||
| + | arr1=np.array(arr*a+b, dtype='float32') | ||
| + | arr1=np.where(arr1<0, 0, arr1) | ||
| + | arr1=np.where(arr1>maks, maks, arr1) | ||
| + | return arr1 | ||
| + | |||
| + | def sig(arr, b, c, maks=255): | ||
| + | arr1=np.array(arr, dtype='float32')-c | ||
| + | #arr1=float(maks)/(1+np.exp(-b*(arr1-c))) | ||
| + | arr1=0.5*(b*arr1/np.sqrt(1+(b*arr1)**2)+1)*maks | ||
| + | return arr1 | ||
| + | |||
| + | class suwaki (object): | ||
| + | def __init__(self, img, maks=255): | ||
| + | self.img=np.array(1.0*(img-np.min(img))*maks/(np.max(img)-np.min(img)), dtype='float32') | ||
| + | self.dtype=img.dtype | ||
| + | #self.b=b | ||
| + | #self.c=c | ||
| + | self.maks=maks | ||
| + | self.widac='' | ||
| + | |||
| + | def pokaz_suw_sig(self): | ||
| + | self.ukryj_suw() | ||
| + | q=np.zeros([50, 200]) | ||
| + | cv2.namedWindow('sigmoida') | ||
| + | cv2.imshow('sigmoida', q) | ||
| + | self.widac='sigmoida' | ||
| + | cv2.createTrackbar('b', 'sigmoida', 10, 20, nothing) | ||
| + | cv2.createTrackbar('c', 'sigmoida', 128, 255, nothing) | ||
| + | |||
| + | def pokaz_suw_lin(self): | ||
| + | self.ukryj_suw() | ||
| + | q=np.zeros([50, 200]) | ||
| + | cv2.namedWindow('liniowy') | ||
| + | cv2.imshow('liniowy', q) | ||
| + | self.widac='liniowy' | ||
| + | cv2.createTrackbar('a', 'liniowy', 10, 20, nothing) | ||
| + | cv2.createTrackbar('x0', 'liniowy', 128, 255, nothing) | ||
| + | |||
| + | def ukryj_suw(self): | ||
| + | if self.widac!='': | ||
| + | cv2.destroyWindow(self.widac) | ||
| + | self.widac='' | ||
| + | |||
| + | def wynik(self): | ||
| + | if self.widac=='sigmoida': | ||
| + | b=2.0**(1.0*cv2.getTrackbarPos('b','sigmoida')-10)*255/self.maks | ||
| + | c=1.0*cv2.getTrackbarPos('c', 'sigmoida')*self.maks/255 | ||
| + | return np.array(sig(self.img, b, c, self.maks), dtype=self.dtype) | ||
| + | elif self.widac=='liniowy': | ||
| + | a=2.0**(cv2.getTrackbarPos('a','liniowy')-10) | ||
| + | b=self.maks/2-a*cv2.getTrackbarPos('x0', 'liniowy')*self.maks/255 | ||
| + | return np.array(lin(self.img, a, b, self.maks), dtype=self.dtype) | ||
| + | else: | ||
| + | return np.array(self.img, dtype=self.dtype) | ||
| + | |||
| + | try: | ||
| + | arr=dataset.pixel_array | ||
| + | |||
| + | if arr.dtype==np.dtype('uint16'): | ||
| + | maks=256*256-1 | ||
| + | else: | ||
| + | maks=255 | ||
| + | cv2.imshow('obraz', arr) | ||
| + | suw=suwaki(arr, maks=maks) | ||
| + | k=-1 | ||
| + | while True: | ||
| + | k=cv2.waitKey(100) | ||
| + | if k==ord('s'): | ||
| + | suw.pokaz_suw_sig() | ||
| + | elif k==ord('l'): | ||
| + | suw.pokaz_suw_lin() | ||
| + | elif k==32: | ||
| + | suw.ukryj_suw() | ||
| + | elif k==27: | ||
| + | break | ||
| + | else: | ||
| + | cv2.imshow('obraz', suw.wynik()) | ||
| + | |||
| + | finally: | ||
| + | cv2.destroyAllWindows() | ||
| + | </source> | ||
Wersja z 14:51, 12 sty 2018
powrót: Programowanie dla fizyków medycznych
Spis treści
Struktura plików DICOM
Prace nad ustandaryzowaniem wymiany danych graficznych pomiędzy urządzeniami medycznymi rozpoczęły się już w 1983 w ramach współpracy American College of Radiology (ACR) i National Electrical Manufactures Association (NEMA). Pierwsza wersja standardu pojawiła się dwa lata później i nosiła nazwę ACR/NEMA 300 V1.0. Wersja ta umożliwiała zapis danych w odpowiednim formacie oraz ujednolicony sposób transferu plików za pomocą sieci lub nośników zewnętrznych. W 1988 roku pojawiła się druga wersja standardu ACR/NEMA V2.0, która opierała się na ujednoliconej terminologii, strukturze informacji odpowiednim kodowaniu danych. Wraz z rozwijaniem standardów pojawiały się nowe zapotrzebowania. Ostatni oficjalna wersja to DICOM 3.0 (Digital Imaging and COmmunications in Medicine) z 1992. Standard ten jest w dalszym ciągu uaktualniany.
Dokumentacja
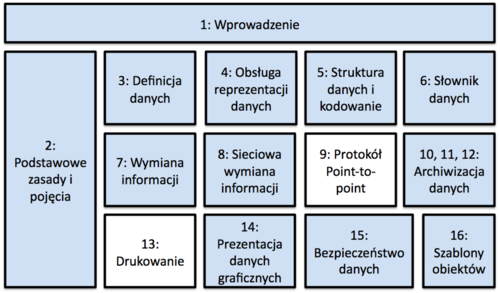
Standard DICOM jest zorganizowany w postaci wieloczęściowego dokumentu (Rys. %i 1) i uaktualniany corocznie w postaci Suplementów.
Obejrzyj stronę domową standardu DICOM: http://dicom.nema.org/, w szczególności: Strategic Document & Principal Contacts
Poszczególne dokumenty są poświęcone strukturze danych, sposobom kodowania, archiwizacji, wymiany informacji oraz aspektom bezpieczeństwa danych. Na zajęciach skupimy się na następujących częściach dokumentacji:
- PS 3.1: Introduction and Overview – wprowadzenie oraz informacje podstawowe na temat standardu
- PS 3.2: Conformance – definicje podstawowych zasad oraz pojęć
- PS 3.3: Information Object Definitions – definicje atrybutów czyli informatyczna reprezentacja fizycznych danych
- PS 3.4: Service Class Specifications – obsługa informatycznych reprezentacji danych
- PS 3.5: Data Structure and Encoding – struktura danych oraz sposób ich kodowania
- PS 3.6: Data Dictionary – spis wszystkich możliwych atrybutów
DICOM-Meta-Information-Header
Dane zawarte w każdym pliku DICOM podzielone są na dwie części:
- część zawierającą informacje o pliku (Dicom-Meta-Information-Header)
- dane jednego obiektu Service-Object Pair Instance (Dicom-Data-Set)
Model informacji określa format danych dla różnych typów informacji, takich jak: obrazy, przebiegi czasowe, obiekty graficzne, raporty, wydruki itp. Dane są grupowane w tematycznych zbiorach (ang. Entities) oraz podzbiorach (ang. Modules). Każdy moduł tworzony jest przez zbiór atrybutów.
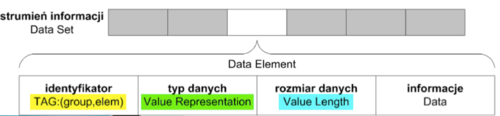
- podstawowa jednostka danych: Data Element
- strumień informacji: Data Set
Data Element stanowi podstawową jednostkę danych, opisywany jest przy pomocy:
- identyfikatora elementu danych (Tag) złożonego z dwóch liczb określających: grupę (Group) oraz element grupy (Element), zapisywanych w postaci liczb heksadecymalnych,
- typu danych (Value Representation), określonego w postaci pary liter w kodzie ASCII i umożliwiającego poprawną interpretację danych,
- rozmiaru elementu (Value Length) wyrażonego w bajtach,
- informacji takich jak: nazwisko pacjenta, rozdzielczość obrazu
Strumień informacyjny (Data Set) jest uporządkowanym strumieniem elementów danych.
Zadanie 1. Na stronie http://northstar-www.dartmouth.edu/doc/idl/html_6.2/DICOM_Attributes.html można znaleźć wygodny opis możliwych pól i odpowiadających im reprezentacji danych. Na tej podstawie znajdź wyjaśnienie następujących oznaczeń:
- (0008,0020): 20120510
- (0008,0030): 101714
- (0008,0060): MG
- (0008,0070): SIEMENS
- (0008,0080): Ovarian Screening, St Barts
- (0010,0010): xxx
- (0010,0040): 0
Rozwiązanie:
- (0008,0020): 20120510 - Study Date
- (0008,0030): 101714 - Study Time
- (0008,0060): MG - Modality
- (0008,0070): SIEMENS - Manufacturer
- (0008,0080): Ovarian Screening, St Barts - Institution Name
- (0010,0010): xxx - Patient's Name
- (0010,0040): 0 - Patient's Sex
Zadanie 2. 56-letnia Janina Kowalska zgłosiła się do Centrum Onkologii z podejrzeniem nowotworu mózgu. 15 lutego 2013 roku zostało wykonanie badanie za pomocą pozytonowej tomografii emisyjnej wykonane na tomografie firmy GE. W czasie badania pacjentka leżała na prawym boku. Za pomocą odpowiednich atrybutów zapisz te informacje w formie pliku DICOM.
Rozwiązanie:
- (0010,0010): Kowalska Janina
- (0010,0040): F
- (0010,1010): 56
- (0008,0080): Centrum Onkologii
- (0008,0020): 20130215
- (0008,0060): PT
- (0008,0070): GE
- (0018,5100): HFDR
Biblioteka PyDICOM
Do obsługi: wczytywania, zmieniania, zapisywania informacji w nagłówku, jak również do pobrania pliku graficznego służy biblioteka pydicom.
Wczytaj plik w formacie DICOM, zawierający skan MRI wraz z informacjami na temat pacjenta i badania. Powstanie w ten sposób obiekt dataset typu pydicom.dataset.FileDataset.
import pydicom as dcm
filename="C:/Users/user/Documents/z_programowanie/DICOM/LADJAN_MRI/localizer - 1/IM-0001-0001.dcm"
dataset = dcm.read_file(filename)
print(dataset)
Obiekt dataset ma strukturę trochę podobną do słownika. Składa się z elementów typu pydicom.dataelem.DataElement. Do elementów można się dostać na różne sposoby: korzystając z nazwy elementu, korzystając z numeru (a ściślej krotki z dwóch numerów wypisywanych zwykle w systemie szesnastkowym).
print(dataset.StudyDate)
print(dataset[(0x8, 0x20)])
element=dataset.data_element('StudyDate')
print(element.value)
Aby dowiedzieć się, jakie elementy wchodzą w skład danego pliku dicomowskiego, można użyć metody dir. Jeśli jako argument poda się string, zostanie wypisana lista tylko tych nazw elementów, które zawierają ten string.
lista1= dataset.dir('pat')
print lista1
for de in lista1:
print(dataset.data_element(de))
Dane o pacjencie mogę pozmieniać lub usunąć.
dataset.StudeDate='20180111' #dzisiejsza data
lista2 = ['PatientAge',
'PatientBirthDate',
'PatientID',
'PatientName',
'PatientPosition',
'PatientSex',
'PatientWeight']
for de in lista2:
if type(dataset.data_element(de).value)==str:
dataset.data_element(de).value='nic'
if 'float' in str(type(dataset.data_element(de).value)):
dataset.data_element(de).value=0
Zmienioną wersję mogę też zapisać (tylko lepiej pod inną nazwą)
dataset.save_as('zmieniony.dcm')
Zadanie 1
Napisz skrypt, który otworzy wszystkie pliki z rozszerzeniem dcm, jakie są w katalogu i usunie z nich informacje osobiste.
Zadanie 2
Zaimportujmy też cv2 i numpy, aby wyświetlić zawartość obrazu.
import cv2
import numpy as np
img=dataset.pixel_array
cv2.imshow('obraz', img)
Zadanie polega na tym, aby za pomocą klawiszy zmieniać jasność wyświetlanego obrazu.
Operacje na obrazie
Przyjmijmy, że wartości pikseli będziemy mnożyć przez funkcję sigmoidalną: [math] f(x)=\frac a {(1+e^{-b(x-c)})} [/math]. Parametr a powinien być równy maksymalnej możliwej wartości piksela, a więc 255 dla obrazów 8-bitowych i 256*256-1=65535 dla obrazów 16-bitowych. Parametr c powinien zawierać się w granicach między 0 i a.
Oprócz tego możemy dokonać innych operacji. Jedną z nich jest progowanie, czyli wyświetlanie jako białych punktów o wartości powyżej progu i jako czarnych wartości poniżej progu. W tym wypadku mielibyśmy tylko jeden parametr (odpowiednik c z funkcji sigmoidalnej).
Inna operacja to mnożenie wartości przez funkcję liniową [math] f(x)=ax+b [/math], dbając o to, by wartości ujemne zostały wyzerowane, a wartości zbyt duże by przyjęły wartość maksymalną. Można do tego napisać własną osobną funkcję, lub wykorzystać funkcję np.where.
Do zmieniania wartości parametrów wykorzystamy suwaki, jakie można utworzyć w okienku obrazu opencv. Metody cv2.createTrackbar oraz cv2.getTrackbarPos pozwolą suwak utworzyć i sprawdzić jego pozycję.
import cv2
import numpy as np
def nothing (x):
pass
img=np.zeros([100, 200], dtype='uint8')
try:
#cv2.namedWindow('suwaczki')
cv2.imshow('suwaczki', img)
cv2.createTrackbar('b','suwaczki',0,255,nothing)
k=-1
while k!=27:
k=cv2.waitKey(0)
b=cv2.getTrackbarPos('b','suwaczki')
print(b)
finally:
cv2.destroyAllWindows()
Zadanie 3
Napisz klasę, która pomoże w manipulacji jasnością i kontrastem obrazu. Powinna mieć następujące własności:
- Przy tworzeniu obiekt powinien dostać przekazaną jako parametr macierz obrazu i ją zapamiętać
- Powinien mieć metody do pokazania i ukrycia osobnego okienka zawierającego suwaki odpowiednie do wybranej metody
- Powinien mieć metodę zwracającą macierz obrazu z dokonanymi na niej operacjami
Główny program powinien wyświetlać obraz, utworzyć dla niego obiekt do operacji i wywoływać odpowiednio jego metody zależnie od klawiszy naciśniętych przez użytkownika.
# -*- coding: utf-8 -*-
"""
Created on Wed Jan 10 16:09:55 2018
@author: user
"""
import pydicom as dcm
import numpy as np
import cv2
filename="C:/Users/user/Documents/z_programowanie/DICOM/LADJAN_MRI/localizer - 1/IM-0001-0001.dcm"
dataset = dcm.dicomio.read_file(filename)
def nothing (x):
pass
def lin(arr, a, b, maks=255):
arr1=np.array(arr*a+b, dtype='float32')
arr1=np.where(arr1<0, 0, arr1)
arr1=np.where(arr1>maks, maks, arr1)
return arr1
def sig(arr, b, c, maks=255):
arr1=np.array(arr, dtype='float32')-c
#arr1=float(maks)/(1+np.exp(-b*(arr1-c)))
arr1=0.5*(b*arr1/np.sqrt(1+(b*arr1)**2)+1)*maks
return arr1
class suwaki (object):
def __init__(self, img, maks=255):
self.img=np.array(1.0*(img-np.min(img))*maks/(np.max(img)-np.min(img)), dtype='float32')
self.dtype=img.dtype
#self.b=b
#self.c=c
self.maks=maks
self.widac=''
def pokaz_suw_sig(self):
self.ukryj_suw()
q=np.zeros([50, 200])
cv2.namedWindow('sigmoida')
cv2.imshow('sigmoida', q)
self.widac='sigmoida'
cv2.createTrackbar('b', 'sigmoida', 10, 20, nothing)
cv2.createTrackbar('c', 'sigmoida', 128, 255, nothing)
def pokaz_suw_lin(self):
self.ukryj_suw()
q=np.zeros([50, 200])
cv2.namedWindow('liniowy')
cv2.imshow('liniowy', q)
self.widac='liniowy'
cv2.createTrackbar('a', 'liniowy', 10, 20, nothing)
cv2.createTrackbar('x0', 'liniowy', 128, 255, nothing)
def ukryj_suw(self):
if self.widac!='':
cv2.destroyWindow(self.widac)
self.widac=''
def wynik(self):
if self.widac=='sigmoida':
b=2.0**(1.0*cv2.getTrackbarPos('b','sigmoida')-10)*255/self.maks
c=1.0*cv2.getTrackbarPos('c', 'sigmoida')*self.maks/255
return np.array(sig(self.img, b, c, self.maks), dtype=self.dtype)
elif self.widac=='liniowy':
a=2.0**(cv2.getTrackbarPos('a','liniowy')-10)
b=self.maks/2-a*cv2.getTrackbarPos('x0', 'liniowy')*self.maks/255
return np.array(lin(self.img, a, b, self.maks), dtype=self.dtype)
else:
return np.array(self.img, dtype=self.dtype)
try:
arr=dataset.pixel_array
if arr.dtype==np.dtype('uint16'):
maks=256*256-1
else:
maks=255
cv2.imshow('obraz', arr)
suw=suwaki(arr, maks=maks)
k=-1
while True:
k=cv2.waitKey(100)
if k==ord('s'):
suw.pokaz_suw_sig()
elif k==ord('l'):
suw.pokaz_suw_lin()
elif k==32:
suw.ukryj_suw()
elif k==27:
break
else:
cv2.imshow('obraz', suw.wynik())
finally:
cv2.destroyAllWindows()