Laboratorium EEG/CSP
Laboratorium_EEG/BSS
Spis treści
Ślepa separacja źródeł
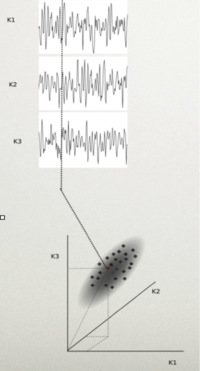
Rozważmy N-kanałowy sygnał EEG. Próbkę tego sygnału możemy przedstawić jako punkt w przestrzeni rozpiętej przez osie, z których każda reprezentuje wartość potencjału w jednym kanale. Cały sygnał tworzy w tej przestrzeni chmurę punktów. Rozciągłość tej chmury w danym kierunku mówi nam o wariancji (zmienności) sygnału w tym kierunku.
Taki zbiór punktów wygodniej jest analizować w układzie współrzędnych zgodnym z osiami głównymi macierzy kowariancji. W dalszej części rozważań założymy, że te przestrzenie, w których rozważamy sygnały są przestrzeniami wektorowymi, a pojedyncze próbki wielokanałowego sygnału są wektorami.
Filtry przestrzenne i ślepa separacja źródeł
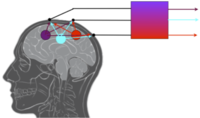
Sygnał EEG jest superpozycją aktywności elektrycznej wielu źródeł. Jak można estymować aktywność samych źródeł?
Niech:
- [math]s(t)[/math] - aktywność niezależnych źródeł,
- [math]x(t)[/math] mierzony sygnał
- [math]A[/math] macierz przejścia taka, że:
- [math]x(t) = A s(t)[/math] (*)
- [math]s(t) = A^{-1}x(t) = P x(t)[/math]
Macierz kowariancji dla sygnałów [math]x(t)[/math] estymujemy tak:
- [math] C_x = E[x(t)x(t)^T][/math]
Podstawiając (*) mamy:
- [math] C_x = E[x x^T] = E[As(As)^T] = A E[s s^T] A^T = A C_s A^T[/math]
Z założenia, że źródła są niezależne wynika, że macierz [math]C_s[/math] jest diagonalna. Przekształcając powyższe równanie możemy zapisać:
- [math]A^{-1} C_x (A^T)^{-1} = P C_x P^T = C_s[/math]
Odwzorowanie [math]P = A^{-1}[/math] diagonalizuje macierz [math]C_x[/math].
Powyższe rozumowanie jest słuszne w przypadku gdy mamy do czynienia z sygnałem stacjonarnym, tzn. jego macierz kowariancji jest niezależna od czasu, czyli przez cały czas aktywna jest ta sama konfiguracja źródeł niezależnych. W przypadku gdy tak nie jest to konstrukcję filtra przestrzennego można oprzeć o jednoczesną diagonalizację macierzy kowariancji odpowiadających różnym stanom osoby badanej.
Common Spatial Pattern
Koncepcja
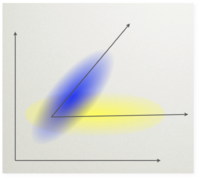
Dla ustalenia uwagi możemy myśleć o eksperymencie wywołującym potencjał P300. Mamy w nim dwie sytuacje eksperymentalne. Oznaczmy ([math]T[/math] - target) próby, w których pojawił się oczekiwany bodziec, zaś ([math]NT[/math] - non-target) gdy pojawił się bodziec standardowy. Chcielibyśmy znaleźć taki montaż, czyli taką kombinację liniową kanałów, które maksymalizuje stosunek mocy (wariancji) sygnałów rejestrowanych w dwóch rożnych warunkach eksperymentalnych.
Formalizm
Metoda ta polega na znalezieniu takiego kierunku [math]w[/math] w przestrzeni sygnałów, że sygnał z warunku [math]T[/math] rzutowany na ten kierunek ma dużą wariancje a sygnał z warunku [math]NT[/math] ma wariancję małą.
Rzutowanie sygnału [math]x(t)[/math] na kierunek [math]w[/math] odbywa się przez policzenie iloczynu skalarnego dla każdej chwili czasu [math]t[/math]:
- [math] s_w(t) = w^T x(t)[/math]
Wariancja tego rzutowanego sygnału to:
- [math] \mathrm{var}(s_w) = E[s_w s_w^T] = E[ w^T x (w^T x)^T] = w^T E[x x^T] w = w^T C_x w [/math]
Zatem znalezienie właściwego kierunku rzutowania można wyrazić jako szukanie maksimum wyrażenia [math] J(w) [/math](jest to tzw. iloraz Rayleigh'a):
- [math]J(w) = \frac{w^T C_T w}{w^T C_{NT} w} [/math]
Ekstremum tego ilorazu można znaleźć poprzez policzenie gradientu [math]J(w)[/math] i przyrównanie go do zera:
- [math] \nabla J(w) = \frac{ 1 C_{T} w+w^T C_{T} 1}{w^T C_{NT} w}-\frac{w^T C_{T} w\left( 1 C_{NT} w+w^T C_{NT} 1\right)}{\left(w^T C_{NT} w\right)^2}[/math]
ponieważ macierze kowariancji są symetryczne
- [math]\nabla J(w) = \frac{ 1}{w^T C_{NT} w}\left[ C_{T} w+ C_{T}w -\frac{w^T C_{T} w}{w^T C_{NT} w} \left( C_{NT} w+ C_{NT}w \right) \right][/math]
- [math]= \frac{ 2}{w^T C_{NT} w}\left[ C_{T}w -\frac{w^T C_{T} w}{w^T C_{NT} w} C_{NT} w \right][/math]
Przyrównując to wyrażenie do zera dostajemy:
- [math] C_{T}w =\frac{w^T C_{T} w}{w^T C_{NT} w} C_{NT} w [/math]
Liczba [math] \lambda = \frac{w^T C_{T} w}{w^T C_{NT} w} C_{NT} w [/math] jest uogólnioną wartością własną, zaś [math]w[/math] jest uogólnionym wektorem własnym odpowiadającym tej wartości.
Aby znaleźć [math] \lambda[/math] i [math]w[/math] wystarczy rozwiązać zagadnienie własne. W matlabie możemy w tym celu wykorzystać funkcję eig
Ćwiczenie symulacyjne
% symulowany eksperyment składa się z sinusoidy udającej alfę spoczynkową i
% funkcji Gaussa udającego potencjał wywołany
% źródła te są symulowane niezależnie a potem mieszane przez macierz L
% symulujemy źródła
% s1 - symuluje alfę
% s2 - symuluje "potencjał wywołany" (ERP)
%ustawiamy parametry do symulacji sygnałów
Fs = 100;
T = 1;
t = 0:1/Fs:T-1/Fs;
N_rep = 100;
N_chan = 2;
s = zeros(N_rep,N_chan, length(t));
X = zeros(N_rep,N_chan, length(t));
% filtr przestrzenny - z takimi wagami trzeba wziąść kanały EEG aby odzyskać sygnały źródłowe
P = [1 2
1.5 1.3];
% topografie - z takimi wagami źródła dokładają się do poszczególnych elektrod
A = P^(-1);
for r =1:N_rep % tworzymy kolejne realizacje "eksperymentu"
s1 = sin(2*pi*11*t +pi/2+ 0*2*pi*rand())+ 0.02*randn(size(t)); % źródło alfa
s2 = exp(-((t-0.8)/0.05).^2)+ 0.01*randn(size(t)); % źródło ERP
s(r,1,:) = s1;
s(r,2,:) = s2;
tmp = squeeze(s(r,:,:));
n = 0*randn(size(tmp));
X(r,:,:) = A*tmp +n; % rzutujemy sygnały źródłowe na elektrody s -> x
end
% wycinamy warunki
% baseline_ind to indeksy pierwszej połowy każdego powtórzenia "baseline"
% ERP_ind to indeksy drugiej połowy każdego powtórzenia zawierająca "ERP"
baseline_ind = find(t<0.5);
ERP_ind = find(t>=0.5);
x_baseline_kan_1 = X(:,1,baseline_ind);
x_baseline_kan_2 = X(:,2,baseline_ind);
x_ERP_kan_1 = X(:,1,ERP_ind);
x_ERP_kan_2 = X(:,2,ERP_ind);
% liczymy średnie macierze kowariancji:
R_E = zeros(N_chan,N_chan);
R_B = zeros(N_chan,N_chan);
for r =1:N_rep
B = squeeze(X(r,:,baseline_ind));
R_B = R_B + B*B' ;
E = squeeze(X(r,:,ERP_ind));
R_E = R_E + E*E' ;
end
R_B = R_B/N_rep;
R_E = R_E/N_rep;
% rozwiązujemy uogólnione zagadnienie własne
[W,Lambda]=eig(R_E,R_B); % możliwa jest też optymalizacja wzg. średniej macierzy kowariancji (R_B+R_A)/2);
% odzyskujemy sygnały źródeł
for r =1:N_rep
S(r,:,:) = W'*squeeze(X(r,:,:));
end
% pobieramy wycinki odpowiadające obu częściom eksperymentu z estymowanych
% źródeł
s_baseline_estymowany_kan1 = squeeze( S(:,1,baseline_ind));
s_baseline_estymowany_kan2 = squeeze( S(:,2,baseline_ind));
s_ERP_estymowany_kan1 = squeeze(S(:,1,ERP_ind));
s_ERP_estymowany_kan2 = squeeze(S(:,2,ERP_ind));
%%%%%%%%%%%%%% Ilustracje %%%%%%%%%%%%%%%%%%%%%%%
% ilustracja sygnałów mierzonych
figure(1);clf
subplot(2,2,1);
plot(t(baseline_ind),(squeeze(X(:,1,baseline_ind)))','b'); hold on
plot(t(ERP_ind),(squeeze( X(:,1,ERP_ind)))','r'); hold off
xlabel('elektroda 1')
title('ilustracja sytuacji pomiarowej -\newline znane są potencjały na elektrodach w dwóch warunkach eksperymentalnych')
subplot(2,2,3);
plot(t(baseline_ind),(squeeze(X(:,1,baseline_ind)))','b'); hold on
plot(t(ERP_ind),(squeeze( X(:,2,ERP_ind)))','r'); hold off
xlabel('elektroda 2')
subplot(1,2,2)
plot(x_baseline_kan_1(:),x_baseline_kan_2(:),'b.');
hold on
plot(x_ERP_kan_1(:),x_ERP_kan_2(:),'r.');
xlim([-2,2])
ylim([-2,2])
axis equal
% wektor własny odpowiadający największej wartości własnej jest
% kierunkiem najbardziej różnicującym warunki eksperymentalne
disp('wartości własne znajdują się na przekątnej macierzy Lambda')
disp(Lambda)
% rysujemy wersory jednostkowe w kierunkach wektorów własnych
w1 = W(:,1);
w1 = w1/norm(w1);
w2 = W(:,2);
w2 = w2/norm(w2);
line([0, w1(1) ],[0,w1(2)],'Color',[0,0.3,0])
text(w1(1),w1(2),'wektor własny 1')
line([0, w2(1) ],[0,w2(2)],'Color',[1,0,1])
text(w2(1),w2(2),'wektor własny 2')
xlabel('Amplituda na elektrodzie 1')
ylabel('Amplituda na elektrodzie 2')
legend('baseline','ERP')
% Ilustracja estymowanych źródeł
figure(2);clf
subplot(2,2,1);
plot(t(baseline_ind),(squeeze(S(:,1,baseline_ind)))','b');hold on
plot(t(ERP_ind),(squeeze(S(:,1,ERP_ind)))','r');hold off
xlabel('estymowane zrodlo 1')
title('ilustracja estymacji -\newline estymowane są potencjały źródeł w dwóch warunkach eksperymentalnych')
subplot(2,2,3);
plot(t(baseline_ind),(squeeze(S(:,2,baseline_ind)))','b');hold on
plot(t(ERP_ind),(squeeze(S(:,2,ERP_ind)))','r');hold off
xlabel('estymowane zrodlo 2');
subplot(1,2,2)
plot(s_baseline_estymowany_kan1(:),s_baseline_estymowany_kan2(:),'b.');
hold on
plot(s_ERP_estymowany_kan1(:),s_ERP_estymowany_kan2(:),'r.');
xlabel('Amplituda estym. źródła 1')
ylabel('Amplituda estym. źródła 2')
legend('baseline','ERP')
Zastosowanie filtra CSP do detekcji potencjału P300
Eksperyment
Przygotowanie do badania:
- założyć czepek z elektrodami w systemie 10-20;
- elektrody referencyjne: M1 i M2;
- elektroda GND w pozycji AFz.
Przygotowanie scenariuszy obci
- w terminalu uruchomić obci srv;
- w terminalu uruchomić obci_gui --preset brain2013;
- w interfejsie GUI zapisujemy scenariusze do własnego katalogu np „P300”
- „P-Brain2013 Signal (with ID)” jako np. „Sygnal”
- „P-Brain2013 Calibration p300” jako „kalibracjaP300”
- „P-Brain 2013 p300” jako „Labirynt”
- w przeglądarce plików otwórz katalog ~/.obci/scenarios/P300. Powinien on zawierać pliki: Sygnal.ini, kalibracjaP300.ini, Labirynt.ini oraz katalogi Sygnal_configs, kalibracjaP300_configs, Labirynt_configs.
- edytujemy parametry peerów.
- z katalogu ~/.obci/scenarios/P300/Sygnal_configs kopiujemy plik amplifier.ini do katalogu ~/.obci/scenarios/P300 jako global_amplifier.ini. To pozwoli nam zmieniać ustawienia wzmacniacza dla wszystkich scenariuszy jednocześnie.
- w naszych scenariuszach zapisanych w plikach Sygnal.ini, kalibracjaP300.ini, Labirynt.ini podmieniamy ścieżkę config = w sekcji [peers.amplifier] tak, aby pokazywała na ten skopiowany plik global_amplifier.ini
- w tym pliku global_amplifier.ini podmieniamy linijki (tak aby były to listy faktycznie wykorzystywanych kanałów) z:
- active_channels
- channel_names
- dodajemy też linijkę: sampling_rate = 256
- wchodzimy po kolei do katalogów: Sygnal_configs, kalibracjaP300_configs, Labirynt_configs i odnajdujemy plik switch_backup.ini. W tym pliku ustawiamy parametr new_scenario na pusty. To spowoduje, że scenariusze te nie będą uruchamiać kolejnych scenariuszy po zakończeniu działania.
- edytujemy plik ~/.obci/scenarios/P300/kalibracjaP300_configs/clasifier.ini
- zmieniamy linię
- edytujemy plik ~/.obci/scenarios/P300/kalibracjaP300_configs/clasifier.ini
- ignore_channels = DriverSaw;AmpSaw;PO7;PO8
- na
- ignore_channels = DriverSaw;AmpSaw;A1;A2
- oraz linię:
- montage_channels = PO7;PO8
- na
- montage_channels = A1;A2
Przeprowadzenie badania:
- Uruchom scenariusz „Sygnał”.
- Tworzy on w katalogu domowym plik o nazwie file_id_name.
- Uruchamiamy Svaroga z terminala poleceniem svarog. W zakładce sygnały on-line odnajdujemy nazwę naszego scenariusza „Sygnal”. Podłączamy się do niego i poprawiamy ewentualnie źle kontaktujące elektrody.
- Jak już jesteśmy zadowoleni z jakości sygnału to zatrzymujemy scenariusz „Sygnal” w obci.
- W pliku file_id_name znajduje się string, który stanowi rdzeń do tworzenia nazw plików, z których korzystają nasze scenariusze. Proszę zmienić ten string np. na: test1.
- Uruchamiamy scenariusz „kalibracjaP300”. Badany będzie oglądał interfejs z trzema literami A B C migającymi w losowej kolejności. Zadaniem jest zliczanie mignięć litery B.
- Po zakończeniu kalibracji uruchamiamy scenariusz „Labirynt”.
- Danych z kalibracji potrzebować będziemy kilka zestawów. Proszę powtórzyć kilkukrotnie scenariusz „kalibracjaP300”. Przed każdym uruchomieniem trzeba zmienić string w pliku file_id_name np. na test??? gdzie ??? oznacza kolejne numery.
Analiza wstępna
- Wczytać dane kalibracyjne do Matlaba i pociąć je na realizacje typu T — „target” (związane z wystąpieniami litery „B”) i NT — „non-target” (pozostałe litery) o długości −200 do +800 ms wokół triggerów. Dla każdej realizacji odjąć trend liniowy.
- Sygnał zmontować wzgl. „połączonych uszu” i wyświetlić średnie przebiegi dla warunku T i NT w układzie topograficznym — wykorzystać w tym celu funkcję topoplot z pakietu Eeglab.
Poniżej zaprezentowany jest przykładowy skrypt do cięcia danych wokół znaczników. Działa on z plikami zawartymi w archiwum:
Korzysta z funkcji pomocniczych dostępnych w dystrybucji obci w katalogu
- /usr/share/openbci/analysis/matlab_obci_signal_processing
Openbci można pobrać z https://github.com/BrainTech/openbci
% ustalamy nzawy plików z danymi
nazwaPliku = 'p_6301423_calibration_p300.obci';
nameOfXMLFile = strcat(nazwaPliku,'.xml');
nameOfTagFile = strcat(nazwaPliku,'.tag'); %tagi = znaczniki zdarzeń
namesOfDataFiles = strcat(nazwaPliku,'.raw');
% inicjujemy obiekt rm
rm = ReadManager(nameOfXMLFile,namesOfDataFiles,nameOfTagFile);
% obieramy przydatne parametry i znaczniki
numberOfChannels = rm.get_param('number_of_channels');
namesOfChannels = rm.get_param('channels_names');
samplingFrequency = rm.get_param('sampling_frequency');
tagsStruct = rm.get_tags();
% tworzenie list znaczników Target i NonTarget
numberOfStruct = length(tagsStruct);
targetTimeStamps = [];
NonTargetTimeStamps = [];
for structNumber = 1:numberOfStruct % iterujemy się przez tagi
if(strcmp(tagsStruct(structNumber).name,'blink')) % szukamy tagów o nazwie 'blink'
index = tagsStruct(structNumber).children.index; % tu jest numer pola stymulacji
target= tagsStruct(structNumber).children.target;% tu jest numer pola na którym wyświetlany jest target
if index == target % warunek na to, że mamy do czynienia z tagiem target
targetTimeStamps = [targetTimeStamps tagsStruct(structNumber).start_timestamp]; %dodajemy timeStamp do listy targetów
else
NonTargetTimeStamps = [NonTargetTimeStamps tagsStruct(structNumber).start_timestamp];%dodajemy timeStamp do listy non-targetów
end
end
end
% pobieramy próbki
samples = double(rm.get_samples()); % konwersja na double jest potrzebna żeby dobrze funkcjonowało filtrowanie
samples=samples(1:8,:); % odrzucamy kanały, które nie mają EEG
numberOfChannels =8;
% filtrujemy dolnoprzepustowo aby odrzucić artefakty sieci i część
% artefaktów mięśniowych
[b,a] = cheby2(6,80,25 /(samplingFrequency/2),'low');
for ch = 1:numberOfChannels
samples(ch,:)=filtfilt(b,a,samples(ch,:));
end
% montujemy dane do wspólnej średniej (common average)
M = -ones(8,8)/8;
M=M+eye(8,8)*9/8;
samples = 0.0715*M*samples;
% wycinamy dane wokół znaczników
PRE = -0.2; % czas przed tagiem w sek.
POST = 0.8; % czas po tagu w sek.
wycinek = floor(PRE*samplingFrequency:POST*samplingFrequency); % tablica ze "standardowymi" indeksami do cięcia
% pobieramy targety
TargetSignal = zeros(length(targetTimeStamps),numberOfChannels, length(wycinek)); % tablica na sygnały target
for trialNumber = 1:length(targetTimeStamps)
trigerOnset = floor(targetTimeStamps(trialNumber)*samplingFrequency);
tenWycinek = wycinek + trigerOnset;
if tenWycinek(1)>0 && tenWycinek(end)<=size(samples,2) % test czy wycinek który chcemy pobrać nie wystaje poza dostępny sygnał
tmpSignal = samples(:,tenWycinek);
tmpSignal = detrend(tmpSignal')'; % usuwanie liniowego trendu - przy krótkich wycinkach działa lepiej niż filtrowanie górnoprzepustowe
TargetSignal(trialNumber, :,:) = tmpSignal;
end
end
% pobieramy non-targety
NonTargetSignal = zeros(length(NonTargetTimeStamps),numberOfChannels, length(wycinek));% tablica na sygnały non-target
for trialNumber = 1:length(NonTargetTimeStamps)
trigerOnset = floor(NonTargetTimeStamps(trialNumber)*samplingFrequency);
tenWycinek = wycinek + trigerOnset;
if tenWycinek(1)>0 && tenWycinek(end)<=size(samples,2)
tmpSignal = samples(:,tenWycinek);
tmpSignal = detrend(tmpSignal')';
NonTargetSignal(trialNumber, :,:) = tmpSignal;
end
end
%
% dla ilustracji podglądamy średnie po powtórzeniach ze wszystkich targetów
% i non-targetów
plot(squeeze(mean(TargetSignal,1))','r');
hold on
plot(squeeze(mean(NonTargetSignal,1))','b')
Analiza CSP
- Korzystając z danych kalibracyjnych wykonać analizę CSP wzmacniającą potencjał P300.
- Zaprezentować średnią ze wszystkich kanałów źródłowych z warunku target (jeden kolor) i non-target (inny kolor) w subplotach ułożonych w prostokątnej siatce. Zaobserwować dla którego kanału średnie różnią się najbardziej. Czy jest związek tego kanału z wartościami własnymi?
- Dla kanału najbardziej różnicującego wykonać mapki topograficzne wektorów odpowiadających:
- filtrowi przestrzennemu
- rzutu topograficznego źródła na elektrody.
- Do wykonania tych mapek wykorzystać funkcję topoplot z pakietu eeglab
- Zbadać powtarzalność topografii pomiędzy plikami konfiguracyjnymi.
Wybór i separacja cech
- Przedstaw na rysunkach nałożone na siebie pojedyncze realizacje z warunków target i non-target po rzutowaniu na wektor [math]w[/math] odpowiadający największej i kolejnej wartości własnej.
- Przedstaw wykresy punktowe, takie że na jednej osi jest moc sygnału odpowiadającego największej wartości własnej a na drugiej osi kolejnej wartości własnej; jeden punkt reprezentuje jedno powtórzenie.
- Wykonaj serię wykresów jak w poprzednim punkcie dla uśrednień kolejno po 2, 4, 6, 8 i 10 realizacjach. Zaobserwuj jak zmienia się separacja w grupach target i non-target.
Filtry przestrzenne dla SSEP
Teoria
Ciekawa koncepcja filtra przestrzennego dla SSVEP zaprezentowana jest tu: http://www.eurasip.org/Proceedings/Eusipco/Eusipco2009/contents/papers/1569193209.pdf
Po krótce można ją rozumieć podobnie do tego co robiliśmy rozważając filtry przestrzenne CSP, z tym, że dla SSVEP oraz innych potencjałów wywołanych stanu ustalonego możemy skorzystać z dodatkowych informacji dotyczących poszukiwanych źródeł. Wiemy mianowicie, że powinny one oscylować z częstością bodźca, i być może jej harmonicznych.
Przyda nam się macierz [math]S[/math] zbudowana tak, że w kolejnych kolumnach znajdują się sinusy i cosinusy kolejnych częstości harmonicznych. Wektory te unormujemy, żeby miały energię równą 1. Innymi słowy macierz [math]S[/math] zbudowana jest z wersorów rozpinających przestrzeń, w której powinien znajdować się sygnał SSVEP.
W matlabie możemy taką macierz zbudować tak:
% Fs - częstość próbkowania
% numberOfSamples - długość sygnału w próbkach
% numberOfHarmonics - liczba harmonicznych, które chcemy włączyć do analizy
t = (0:1:numberOfSamples - 1)/Fs;
S = zeros(numberOfSamples, 2*numberOfHarmonics);
for harmonicNumber = 1:numberOfHarmonics
c = cos(2*pi*stimulationFrequency*harmonicNumber*t);
s = sin(2*pi*stimulationFrequency*harmonicNumber*t);
S(:,(harmonicNumber - 1)*2 + 1) = c/norm(c);
S(:,(harmonicNumber - 1)*2 + 2) = s/norm(s);
end
Aby w badanym sygnale znaleźć składowe odpowiadające SSVEP musimy rzutować sygnał [math]X[/math] (macierz sygnałów kanały x próbki) na przestrzeń rozpiętą przez [math]S[/math]:
- [math]A = X*S[/math]
Macierz [math]A[/math] zawiera współczynniki będące iloczynami skalarnymi sygnałów i wersorów. Mówią one o tym "jak duże" jest sinusa bądź cosinusa o danej częstości w pierwotnym sygnale. Komponenty SSVEP zawarte w sygnale [math]X[/math] odzyskujemy tak:
- [math]\mathrm{SSVEP} = A S^T[/math]
Modelujemy rejestrowany sygnał jako:
- [math]X = \mathrm{SSVEP} + Y [/math]
gdzie:
- [math]Y = X-\mathrm{SSVEP}[/math]
- to wszystkie komponenty sygnału, które nas nie interesują.
Filtr przestrzenny, który chcemy zbudować powinien maksymalizować stosunek wariancji [math]\mathrm{SSVEP} = A S^T[/math] do wariancji [math]Y = X-\mathrm{SSVEP}[/math]. Dalej możemy zastosować technikę znaną z konstrukcji filtrów CSP, tzn. maksymalizacji ilorazu Rayleigh'a za pomocą rozwiązania ogólnionego zagadnienia własnego dla macierzy kowariancji [math]\mathrm{SSVEP} [/math] i [math]Y [/math].
Poniżej prosta demonstracja dla danych zebranych EEG przy stymulacji SSVEP z częstotliwością 38Hz.
Spakowane dane: Plik:PrzykladoweDaneSSVEP.mat.gz. W oparciu o powyższy opis proszę zaimplementować funkcję cosSinCSP. Prawidłowo zaimplementowana funkcja wraz z poniższym kodem powinna generować rysunek:
% wczytujemy dane
load('przykladoweDaneSSVEP.mat');
% numberOfChannels numberOfSamples numberOfTrials
P = zeros(numberOfChannels,numberOfChannels);
numberOfHarmonics = 3;
signal = X.data; % ( powtórzenie, kanał, próbki)
S = zeros(size(signal));
P = cosSinCSP(signal,X.stimulation,numberOfHarmonics,X.sampling);
for powt = 1:size(signal,1)
S(powt,:,:) = P'*squeeze(signal(powt,:,:));
end
figure('Name',['Stymulacja: ',num2str(X.stimulation),' Hz'])
for i =1:numberOfChannels
% rysujemy widma uśrednione po realizacjach dla danych
% z oryginalnych kanałów EEG
subplot(2,8,i)
PP=0;
for rep = 1:freq_trial_count
x = signal(rep,i,:);
[Pxx,ff]=pwelch(x, X.sampling, 1, X.sampling, X.sampling);
PP =PP + Pxx;
end
plot(ff(ff<60),PP(ff<60))
title(namesOfChannels{i})
% rysujemy widma uśrednione po realizacjach dla danych
% z estymowanych źródeł CSP
subplot(2,8,8+i)
PP=0;
for rep = 1:freq_trial_count
s = S(rep,i,:);
[Pss,ff]=pwelch(s, X.sampling, 1, X.sampling, X.sampling);
PP =PP + Pss;
end
plot(ff(ff<60),PP(ff<60))
title(['źródło CSP: ', num2str(i)])
end
Przykładowy skrypt i dane prezentujący konstrukcję i działanie tego typu filtrów przestrzennych dla pełnych danych z eksperymentu SSVEP: Plik:SSVEP demo csp.tar.gz
ICA jako filtr przestrzenny
Dane do tej części ćwiczeń proszę pobrać i rozpakować w swoim katalogu. ... Pochodzą one z eksperymentu, w którym osoba badana siedziała z zamkniętymi oczami słuchając nagrania czytanego spokojnym głosem. Metadane opisujące sygnał znajdują się w pliku Miro.xml, zaś lokalizacje elektrod w pliku Miro-10-20-Cap.locs.
Proszę:
- wczytać dane do eeglaba
- wyedytować lokalizację elektrod
- usunąć kanały nie zawierające EEG
- zmienić referencje na średnią z kanałów A1 i A2
- przefiltrować filtrem FIR górnoprzepustowym z częstością odcięcia 0,5 Hz
- obejrzeć wstępnie przygotowane dane
- policzyć ICA na całym sygnale
- obejrzeć właściwości otrzymanych komponentów
- Czy są wśród nich takie, które zawierają znaczny udział rytmu alfa?
- Jaka jest ich topografia?
- usunąć wszystkie komponenty nie zawierające alfy
- odtworzyć z tych komponentów sygnał na elektrodach