Nowe technologie w fizyce biomedycznej/Kamery 3D: Różnice pomiędzy wersjami
| (Nie pokazano 15 pośrednich wersji utworzonych przez tego samego użytkownika) | |||
| Linia 12: | Linia 12: | ||
**W ramach pomiarów studenci wykonają standardowy test wykorzystywany w medycynie sportowej do oceny prawdopodobieństwa urazów więzadła krzyżowego przedniego (ACL). | **W ramach pomiarów studenci wykonają standardowy test wykorzystywany w medycynie sportowej do oceny prawdopodobieństwa urazów więzadła krzyżowego przedniego (ACL). | ||
| − | [[Media:3D_Kinect.pdf]] Informacje wstępne oraz opis zadań | + | [[Media:3D_Kinect.pdf]] Informacje wstępne oraz opis zadań <br> |
| + | [[Media:3D_Kinect_ENG.pdf]] Theoretical information and description of tasks | ||
Zajęcia 2,3,4: | Zajęcia 2,3,4: | ||
| Linia 37: | Linia 38: | ||
==Pomiary== | ==Pomiary== | ||
| + | Pomiary przeprowadzane są w środowisku OpenBCI przy użyciu aplikacji Brain4edu. Obsługa sensora wraz z algorytmami estymacji poszczególnych pozycji anatomicznych śledzonej postaci, możliwa jest dzięki bibliotekom OpenNI i NiTE. | ||
| + | |||
| + | Zaczynamy od uruchomienia aplikacji Brain. W prawym górnym rogu ekranu pojawi się ikona mózgu będąca interfejsem graficznym aplikacji. Do pomiarów przy użyciu kamery 3D wybieramy opcję 'Kinect App', która otworzy okno do zapisu lub odczytu danych. | ||
| + | |||
| + | [[Plik:KinectApp.png|300px|thumb|left|<figure id="fig:wbb"></figure>Interfejs graficzny aplikacji Brain4edu]] | ||
| + | [[Plik:KinectAppStart.png|300px|thumb|right|<figure id="fig:wbb"></figure>Okno wyboru rejestracji/odczytu danych aplikacji KinectApp]] | ||
| + | |||
| + | W wyświetlonym oknie wpisz katalog zapisu/odczytu, wybierz tryb działania, a następnie naciśnij przycisk Start. Po uruchomieniu wyświetlone zostanie okno z podglądem zarówno mapy głębokości jak i obrazu RGB. | ||
| + | |||
| + | <!-- | ||
Rejestracja danych odbywa się w środowisku OpenBCI. System ten uruchamiamy wykonując w terminalu polecenie: | Rejestracja danych odbywa się w środowisku OpenBCI. System ten uruchamiamy wykonując w terminalu polecenie: | ||
| Linia 58: | Linia 69: | ||
[[Plik:obci_gui.png|600px|thumb|center|<figure id="fig:obci_kinect"></figure>Scenariusz w OpenBCI do rejestracji danych przy pomocy Kinecta.]] | [[Plik:obci_gui.png|600px|thumb|center|<figure id="fig:obci_kinect"></figure>Scenariusz w OpenBCI do rejestracji danych przy pomocy Kinecta.]] | ||
| + | --> | ||
=== Drop Vertical Jump === | === Drop Vertical Jump === | ||
| Linia 69: | Linia 81: | ||
Zeskok z pionowym wyskokiem powtórzony został 3-krotnie w celu większej wiarygodności wyników. Należy zatem dla każdego powtórzenia oddzielnie wyznaczyć momenty IC oraz PF, wyliczyć wartość każdego z parametrów. A następnie należy uśrednić uzyskane wyniki i podać je wraz z niepewnością średniej. Omawiane wskaźniki biomechaniczne: | Zeskok z pionowym wyskokiem powtórzony został 3-krotnie w celu większej wiarygodności wyników. Należy zatem dla każdego powtórzenia oddzielnie wyznaczyć momenty IC oraz PF, wyliczyć wartość każdego z parametrów. A następnie należy uśrednić uzyskane wyniki i podać je wraz z niepewnością średniej. Omawiane wskaźniki biomechaniczne: | ||
| + | *knee valgus motion (KVM) - przesunięcie kolan w płaszczyźnie czołowej (x) pomiędzy momentami IC a PF | ||
[[Plik:KVM.png|400px|thumb|center|<figure id="fig:kinect_joints"></figure>Knee valgus motion]] | [[Plik:KVM.png|400px|thumb|center|<figure id="fig:kinect_joints"></figure>Knee valgus motion]] | ||
| − | *frontal plane knee angle (FPKA) w dwóch momentach | + | *frontal plane knee angle (FPKA) - kąt pod jakim znajduje się wektor (od kolana do stopy) oddzielnie dla prawej i lewej nogi, w dwóch momentach: IC oraz PF |
[[Plik:FPKA.png|400px|thumb|center|<figure id="fig:kinect_joints"></figure>Frontal plane knee angle]] | [[Plik:FPKA.png|400px|thumb|center|<figure id="fig:kinect_joints"></figure>Frontal plane knee angle]] | ||
| − | *knee-to-ankle separation ration (KASR) w momencie PF | + | *knee-to-ankle separation ration (KASR) - stosunek rozsunięcia kolan do kostek w momencie PF |
[[Plik:KASR.png|400px|thumb|center|<figure id="fig:kinect_joints"></figure>knee-to-ankle separation ration]] | [[Plik:KASR.png|400px|thumb|center|<figure id="fig:kinect_joints"></figure>knee-to-ankle separation ration]] | ||
| − | *zmiana w czasie średniej pozycji bioder. | + | *zmiana w czasie średniej pozycji bioder, kolan i stóp. |
| − | Wyniki opracowane powinny zostać w formie tabeli, gdzie zestawione zostaną wartości KVM, FPKA, KASR otrzymane dla każdej osoby z grupy (uśrednione po realizacjach - 3 powtórzeniach). Trajektoria średniej pozycji bioder podczas wykonywania zadania powinna zostać przedstawiona na wykresie. | + | Wyniki opracowane powinny zostać w formie tabeli, gdzie zestawione zostaną wartości KVM, FPKA, KASR otrzymane dla każdej osoby z grupy (uśrednione po realizacjach - 3 powtórzeniach). Trajektoria (w kierunku pionowym i poziomym) średniej pozycji bioder, kolan i stóp podczas wykonywania zadania powinna zostać przedstawiona na wykresie. |
===Analiza danych=== | ===Analiza danych=== | ||
| − | + | Implementacja klasy Serialization umożliwiająca łatwe wczytanie danych (proszę zapisać ten plik jako KinectUtils). | |
| + | |||
| + | {{Solution|title=implementacja klasy|text= | ||
<source lang="Python"> | <source lang="Python"> | ||
| − | #!/usr/bin/env | + | #!/usr/bin/env python3 |
# -*- coding: utf-8 -*- | # -*- coding: utf-8 -*- | ||
| − | + | import struct | |
| − | + | ||
| − | + | class KinectException(Exception): | |
| − | + | pass | |
| + | |||
| + | class JointStruct(struct.Struct): | ||
| + | def __init__(self): | ||
| + | super(JointStruct, self).__init__('iffffff') | ||
| + | |||
| + | class HandStruct(struct.Struct): | ||
| + | def __init__(self): | ||
| + | super(HandStruct, self).__init__('ifffff') | ||
| + | |||
| + | class UserStruct(struct.Struct): | ||
| + | def __init__(self): | ||
| + | super(UserStruct, self).__init__('hfffi') | ||
| + | |||
| + | class FrameStruct(struct.Struct): | ||
| + | def __init__(self): | ||
| + | super(FrameStruct, self).__init__('iQ') | ||
| + | |||
| + | class HeaderStruct(struct.Struct): | ||
| + | def __init__(self): | ||
| + | super(HeaderStruct, self).__init__('i??diQiQ') | ||
| + | |||
| + | class Point3Mock(object): | ||
| + | def __init__(self, x, y, z): | ||
| + | self.x = x | ||
| + | self.y = y | ||
| + | self.z = z | ||
| + | |||
| + | class JointMock(object): | ||
| + | def __init__(self, id, positionConfidence, x, y, z, x_converted, y_converted): | ||
| + | self.id = id | ||
| + | self.positionConfidence = positionConfidence | ||
| + | self.x = x | ||
| + | self.y = y | ||
| + | self.z = z | ||
| + | self.x_converted = x_converted | ||
| + | self.y_converted = y_converted | ||
| + | |||
| + | class SkeletonFrameMock(object): | ||
| + | def __init__(self, frame_index, frame_timestamp, user_id, x, y, z, user_state, joints): | ||
| + | self.frame_index = frame_index | ||
| + | self.frame_timestamp = frame_timestamp | ||
| + | self.user_id = user_id | ||
| + | self.user_x = x | ||
| + | self.user_y = y | ||
| + | self.user_z = z | ||
| + | self.user_state = user_state | ||
| + | self.joints = joints | ||
| + | |||
| + | class HandDataMock(object): | ||
| + | def __init__(self, hand_id, x, y, z, x_converted, y_converted): | ||
| + | self.id = hand_id | ||
| + | self.x = x | ||
| + | self.y = y | ||
| + | self.z = z | ||
| + | self.x_converted = x_converted | ||
| + | self.y_converted = y_converted | ||
| + | |||
| + | class HandFrameMock(object): | ||
| + | def __init__(self, frame_index, frame_timestamp, hands): | ||
| + | self.frame_index = frame_index | ||
| + | self.frame_timestamp = frame_timestamp | ||
| + | self.hands = hands | ||
| + | |||
| + | class Serialization(object): | ||
| + | def __init__(self): | ||
| + | self.joint_s = JointStruct() | ||
| + | self.hand_s = HandStruct() | ||
| + | self.user_s = UserStruct() | ||
| + | self.frame_s = FrameStruct() | ||
| + | self.header_s = HeaderStruct() | ||
| + | |||
| + | def serialize_frame(self, header, hand_frame, user_frame, frame_index): | ||
| + | buf = self.header_s.pack(*header) | ||
| + | buf += self.serialize_skeleton(user_frame, frame_index) | ||
| + | buf += self.serialize_hands(hand_frame, frame_index) | ||
| + | return buf | ||
| + | |||
| + | def unserialize_frame(self, data_file): | ||
| + | data = [] | ||
| + | try: | ||
| + | data += self.header_s.unpack(data_file.read(self.header_s.size)) | ||
| + | data.append(self.unserialize_skeleton(data_file)) | ||
| + | data.append(self.unserialize_hands(data_file)) | ||
| + | except struct.error: | ||
| + | return | ||
| + | return data | ||
| + | |||
| + | def register_hand_coordinates(self, convert_hand_coordinates): | ||
| + | self.convert_hand_coordinates = convert_hand_coordinates | ||
| + | |||
| + | def register_joint_coordinates(self, convert_joint_coordinates): | ||
| + | self.convert_joint_coordinates = convert_joint_coordinates | ||
| + | |||
| + | def serialize_hands(self, hand_frame, frame_index): | ||
| + | if hand_frame is not None: | ||
| + | buf = self.frame_s.pack(frame_index, hand_frame.timestamp) | ||
| + | hands = hand_frame.hands | ||
| + | if hands: | ||
| + | if len(hands) == 1: | ||
| + | hand = hands[0] | ||
| + | if hand.isTracking(): | ||
| + | rc, x_new, y_new = self.convert_hand_coordinates(hand.position.x, hand.position.y, hand.position.z) | ||
| + | buf += self.hand_s.pack(hand.id, | ||
| + | hand.position.x, | ||
| + | hand.position.y, | ||
| + | hand.position.z, | ||
| + | x_new, | ||
| + | y_new) | ||
| + | else: | ||
| + | buf += self.hand_s.pack(0, 0, 0, 0, 0, 0) | ||
| + | buf += self.hand_s.pack(0, 0, 0, 0, 0, 0) | ||
| + | else: | ||
| + | for hand in hands[:2]: | ||
| + | rc, x_new, y_new = self.convert_hand_coordinates(hand.position.x, hand.position.y, hand.position.z) | ||
| + | buf += self.hand_s.pack(hand.id, | ||
| + | hand.position.x, | ||
| + | hand.position.y, | ||
| + | hand.position.z, | ||
| + | x_new, | ||
| + | y_new) | ||
| + | else: | ||
| + | for i in range(2): | ||
| + | buf += self.hand_s.pack(0, 0, 0, 0, 0, 0) | ||
| + | else: | ||
| + | buf = self.frame_s.pack(0, 0) | ||
| + | for i in range(2): | ||
| + | buf += self.hand_s.pack(0, 0, 0, 0, 0, 0) | ||
| + | return buf | ||
| + | |||
| + | def serialize_joint(self, joint): | ||
| + | if joint is not None: | ||
| + | pos = joint.position | ||
| + | rc, x_new, y_new = self.convert_joint_coordinates(pos.x, pos.y, pos.z) | ||
| + | return self.joint_s.pack(joint.type, | ||
| + | joint.positionConfidence, | ||
| + | joint.position.x, | ||
| + | joint.position.y, | ||
| + | joint.position.z, | ||
| + | x_new, | ||
| + | y_new) | ||
| + | else: | ||
| + | return self.joint_s.pack(0, 0, 0, 0, 0, 0, 0) | ||
| + | |||
| + | def serialize_skeleton(self, user_frame, frame_index): | ||
| + | if user_frame is not None: | ||
| + | buf = self.frame_s.pack(frame_index, user_frame.timestamp) | ||
| + | users = user_frame.users | ||
| + | if users: | ||
| + | user = users[0] | ||
| + | user_coordinates = user.centerOfMass | ||
| + | state = int(user.skeleton.state) | ||
| + | buf += self.user_s.pack(user.id, | ||
| + | user_coordinates.x, | ||
| + | user_coordinates.y, | ||
| + | user_coordinates.z, | ||
| + | state) | ||
| + | for i in range(15): | ||
| + | buf += self.serialize_joint(user.skeleton[JointType(i)]) | ||
| + | else: | ||
| + | buf += self.user_s.pack(0, 0, 0, 0, 0) | ||
| + | for i in range(15): | ||
| + | buf += self.serialize_joint(None) | ||
| + | else: | ||
| + | buf = self.frame_s.pack(0, 0) | ||
| + | buf += self.user_s.pack(0, 0, 0, 0, 0) | ||
| + | for i in range(15): | ||
| + | buf += self.serialize_joint(None) | ||
| + | return buf | ||
| + | |||
| + | def unserialize_hands(self, f): | ||
| + | v = self.frame_s.unpack(f.read(self.frame_s.size)) | ||
| + | data = [] | ||
| + | for i in range(2): | ||
| + | hands = self.hand_s.unpack(f.read(self.hand_s.size)) | ||
| + | data.append(HandDataMock(hands[0], hands[1], hands[2], hands[3], hands[4], hands[5])) | ||
| + | return HandFrameMock(v[0], v[1], data) | ||
| + | |||
| + | def unserialize_joint(self, f): | ||
| + | v = self.joint_s.unpack(f.read(self.joint_s.size)) | ||
| + | return JointMock(*v) | ||
| + | |||
| + | def unserialize_skeleton(self, f): | ||
| + | v = [] | ||
| + | v += self.frame_s.unpack(f.read(self.frame_s.size)) | ||
| + | v += self.user_s.unpack(f.read(self.user_s.size)) | ||
| + | joints = [] | ||
| + | for i in range(15): | ||
| + | joints.append(self.unserialize_joint(f)) | ||
| + | return SkeletonFrameMock(v[0], v[1], v[2], v[3], v[4], v[5], v[6], joints) | ||
class KinectDataReader(object): | class KinectDataReader(object): | ||
| Linia 99: | Linia 303: | ||
def readNextFrame(self): | def readNextFrame(self): | ||
return self._s.unserialize_frame(self.in_algs_file) | return self._s.unserialize_frame(self.in_algs_file) | ||
| + | |||
| + | </source> | ||
| + | {{clear}} | ||
| + | }} | ||
| + | |||
| + | Przykładowy skrypt do wczytywania wyników algorytmów śledzenia pozycji anatomicznych (wraz z metadanymi rejestracji): | ||
| + | |||
| + | <source lang="Python"> | ||
| + | #!/usr/bin/env python3 | ||
| + | # -*- coding: utf-8 -*- | ||
| + | |||
| + | from KinectUtils import KinectDataReader | ||
| + | |||
| + | FILE_PATH = '......' | ||
| + | file_name = '......' | ||
| + | |||
| + | kinect = KinectDataReader(FILE_PATH+file_name) | ||
first_time = 0 | first_time = 0 | ||
| Linia 104: | Linia 325: | ||
Time = [] | Time = [] | ||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
while True: | while True: | ||
frame = kinect.readNextFrame() | frame = kinect.readNextFrame() | ||
Aktualna wersja na dzień 06:48, 9 maj 2018
Spis treści
Kamery 3D
Plan zajęć
Zajęcia 1:
- Wstęp teoretyczny:
- Budowa i zasada działania sensora Kinect.
- Opis podstawowych bibliotek wykorzystywanych do komunikacji z urządzeniem.
- Profesjonalne systemy do rejestracji ruchu
- Zastosowania w biomechanice oraz rehabilitacji.
- Zapoznanie się z działaniem sensora Kinect
- Pomiary:
- W ramach pomiarów studenci wykonają standardowy test wykorzystywany w medycynie sportowej do oceny prawdopodobieństwa urazów więzadła krzyżowego przedniego (ACL).
Media:3D_Kinect.pdf Informacje wstępne oraz opis zadań
Media:3D_Kinect_ENG.pdf Theoretical information and description of tasks
Zajęcia 2,3,4:
- Analiza zebranych danych
- Zaprezentowanie uzyskanych wyników
Sensor Kinect
Śledzenie ruchów postaci ludzkiej w ogólności znajduje zastosowanie m.in. w analizie chodu, diagnostyce chorób związanych z układem ruchu człowieka, analizie ruchu sportowców, czy nawet w procesie animacji postaci na potrzeby produkcji filmów i gier. W tym celu wykorzystuje się głównie profesjonalne markerowe systemy śledzenia ruchu (marker-based motion capture systems) określane w skrócie jako systemy mocap. Systemy te wymagają zastosowania specjalistycznego sprzętu, a na ciele śledzonej postaci muszą zostać umieszczone odpowiednie markery, przez co rejestracja musi odbywać się w warunkach laboratoryjnych. Systemy te nie nadają się do śledzenia ruchu w czasie rzeczywistym, a przesłonięcie lub przemieszczenie markerów w trakcie ruchu może być przyczyną błędów. Z tych względów coraz większe zainteresowanie zyskują bezmarkerowe systemy śledzenia ruchu, które wykorzystują zaawansowane algorytmy analizy obrazu.
Sensor Kinect firmy Microsoft do śledzenia ruchu postaci wykorzystuje informacje z kamery głębokości. Znajduje on zastosowanie w różnego rodzaju systemach umożliwiających interakcję człowiek-komputer, ale zaczyna również budzić coraz większe zainteresowanie w dziedzinach związanych z biomechaniką i rehabilitacją. W tym celu konieczne jest jednak określenie dokładności pozycji estymowanych przy pomocy bibliotek obsługujących sensor. Problem ten został poruszony w pracy (Webster, 2014).
Budowa sensora Kinect Xbox 360 (rys. 1):
- kamera wizyjna RGB (typu CMOS, o rozdzielczości 640x480) - przesyła serię obrazów z prędkością 30 klatek na sekundę,
- kamera głębokości (typu CMOS, o rozdzielczości ~300x200) - zwraca informację o głębokości poprzez analizę zniekształconej przez obiekt wiązki promieni podczerwonych,
- emiter podczerwieni - emituje wiązkę promieni podczerwonych,
- 4 mikrofony kierunkowe - wykorzystywane przez funkcje rozpoznawania mowy,
- napęd umożliwiający ruch głowicą z akcelerometrem.
Pomiary
Pomiary przeprowadzane są w środowisku OpenBCI przy użyciu aplikacji Brain4edu. Obsługa sensora wraz z algorytmami estymacji poszczególnych pozycji anatomicznych śledzonej postaci, możliwa jest dzięki bibliotekom OpenNI i NiTE.
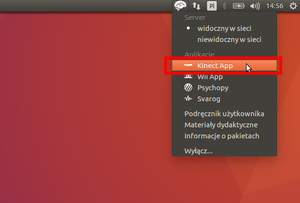
Zaczynamy od uruchomienia aplikacji Brain. W prawym górnym rogu ekranu pojawi się ikona mózgu będąca interfejsem graficznym aplikacji. Do pomiarów przy użyciu kamery 3D wybieramy opcję 'Kinect App', która otworzy okno do zapisu lub odczytu danych.
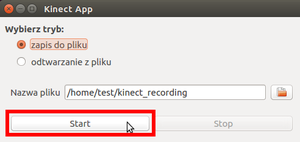
W wyświetlonym oknie wpisz katalog zapisu/odczytu, wybierz tryb działania, a następnie naciśnij przycisk Start. Po uruchomieniu wyświetlone zostanie okno z podglądem zarówno mapy głębokości jak i obrazu RGB.
Drop Vertical Jump
Zeskok z następującym wyskokiem pionowym (drop vertical jump, DVJ) należy do badań przesiewowych, pozwalających oszacować ryzyko występienia urazu (w szczególności więzadła krzyżowego przedniego u kobiet) lub określić efekty rehabilitacji. Przed rozpoczęciem skoku osoba badana stoi na platformie o wysokości ok. 30 cm. Ma ona za zadanie wykonać zeskok z platformy na ziemię, a następnie maksymalny skok pionowy w górę. W dalszej analizie interesujące będą momenty: kontaktu pięt z podłożem (initial contact, IC) zaraz po wykonaniu zeskoku oraz moment maksymalnego zgięcia kolan (peak flexion, PF) zaraz po IC, ale przed oderwaniem pięt od podłoża w celu wykonania skoku pionowego.
Analiza danych
Studenci zapoznają się z modułami (napisanymi w języku programowania Python) do wczytywania oraz wstępnego przetwarzania danych. Następnie samodzielnie wyznaczą wskaźniki biomechaniczne, opisane w pracy (Stone et al., 2013), którą można znaleźć pod adresem: [1].
Zeskok z pionowym wyskokiem powtórzony został 3-krotnie w celu większej wiarygodności wyników. Należy zatem dla każdego powtórzenia oddzielnie wyznaczyć momenty IC oraz PF, wyliczyć wartość każdego z parametrów. A następnie należy uśrednić uzyskane wyniki i podać je wraz z niepewnością średniej. Omawiane wskaźniki biomechaniczne:
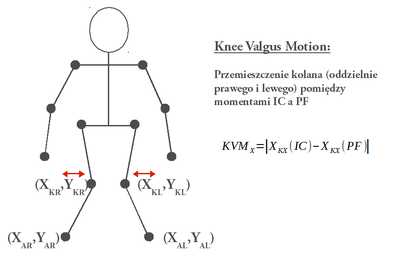
- knee valgus motion (KVM) - przesunięcie kolan w płaszczyźnie czołowej (x) pomiędzy momentami IC a PF
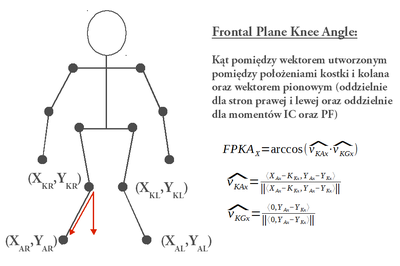
- frontal plane knee angle (FPKA) - kąt pod jakim znajduje się wektor (od kolana do stopy) oddzielnie dla prawej i lewej nogi, w dwóch momentach: IC oraz PF
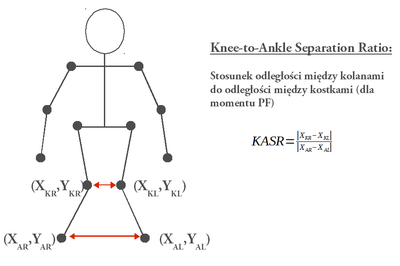
- knee-to-ankle separation ration (KASR) - stosunek rozsunięcia kolan do kostek w momencie PF
- zmiana w czasie średniej pozycji bioder, kolan i stóp.
Wyniki opracowane powinny zostać w formie tabeli, gdzie zestawione zostaną wartości KVM, FPKA, KASR otrzymane dla każdej osoby z grupy (uśrednione po realizacjach - 3 powtórzeniach). Trajektoria (w kierunku pionowym i poziomym) średniej pozycji bioder, kolan i stóp podczas wykonywania zadania powinna zostać przedstawiona na wykresie.
Analiza danych
Implementacja klasy Serialization umożliwiająca łatwe wczytanie danych (proszę zapisać ten plik jako KinectUtils).
#!/usr/bin/env python3
# -*- coding: utf-8 -*-
import struct
class KinectException(Exception):
pass
class JointStruct(struct.Struct):
def __init__(self):
super(JointStruct, self).__init__('iffffff')
class HandStruct(struct.Struct):
def __init__(self):
super(HandStruct, self).__init__('ifffff')
class UserStruct(struct.Struct):
def __init__(self):
super(UserStruct, self).__init__('hfffi')
class FrameStruct(struct.Struct):
def __init__(self):
super(FrameStruct, self).__init__('iQ')
class HeaderStruct(struct.Struct):
def __init__(self):
super(HeaderStruct, self).__init__('i??diQiQ')
class Point3Mock(object):
def __init__(self, x, y, z):
self.x = x
self.y = y
self.z = z
class JointMock(object):
def __init__(self, id, positionConfidence, x, y, z, x_converted, y_converted):
self.id = id
self.positionConfidence = positionConfidence
self.x = x
self.y = y
self.z = z
self.x_converted = x_converted
self.y_converted = y_converted
class SkeletonFrameMock(object):
def __init__(self, frame_index, frame_timestamp, user_id, x, y, z, user_state, joints):
self.frame_index = frame_index
self.frame_timestamp = frame_timestamp
self.user_id = user_id
self.user_x = x
self.user_y = y
self.user_z = z
self.user_state = user_state
self.joints = joints
class HandDataMock(object):
def __init__(self, hand_id, x, y, z, x_converted, y_converted):
self.id = hand_id
self.x = x
self.y = y
self.z = z
self.x_converted = x_converted
self.y_converted = y_converted
class HandFrameMock(object):
def __init__(self, frame_index, frame_timestamp, hands):
self.frame_index = frame_index
self.frame_timestamp = frame_timestamp
self.hands = hands
class Serialization(object):
def __init__(self):
self.joint_s = JointStruct()
self.hand_s = HandStruct()
self.user_s = UserStruct()
self.frame_s = FrameStruct()
self.header_s = HeaderStruct()
def serialize_frame(self, header, hand_frame, user_frame, frame_index):
buf = self.header_s.pack(*header)
buf += self.serialize_skeleton(user_frame, frame_index)
buf += self.serialize_hands(hand_frame, frame_index)
return buf
def unserialize_frame(self, data_file):
data = []
try:
data += self.header_s.unpack(data_file.read(self.header_s.size))
data.append(self.unserialize_skeleton(data_file))
data.append(self.unserialize_hands(data_file))
except struct.error:
return
return data
def register_hand_coordinates(self, convert_hand_coordinates):
self.convert_hand_coordinates = convert_hand_coordinates
def register_joint_coordinates(self, convert_joint_coordinates):
self.convert_joint_coordinates = convert_joint_coordinates
def serialize_hands(self, hand_frame, frame_index):
if hand_frame is not None:
buf = self.frame_s.pack(frame_index, hand_frame.timestamp)
hands = hand_frame.hands
if hands:
if len(hands) == 1:
hand = hands[0]
if hand.isTracking():
rc, x_new, y_new = self.convert_hand_coordinates(hand.position.x, hand.position.y, hand.position.z)
buf += self.hand_s.pack(hand.id,
hand.position.x,
hand.position.y,
hand.position.z,
x_new,
y_new)
else:
buf += self.hand_s.pack(0, 0, 0, 0, 0, 0)
buf += self.hand_s.pack(0, 0, 0, 0, 0, 0)
else:
for hand in hands[:2]:
rc, x_new, y_new = self.convert_hand_coordinates(hand.position.x, hand.position.y, hand.position.z)
buf += self.hand_s.pack(hand.id,
hand.position.x,
hand.position.y,
hand.position.z,
x_new,
y_new)
else:
for i in range(2):
buf += self.hand_s.pack(0, 0, 0, 0, 0, 0)
else:
buf = self.frame_s.pack(0, 0)
for i in range(2):
buf += self.hand_s.pack(0, 0, 0, 0, 0, 0)
return buf
def serialize_joint(self, joint):
if joint is not None:
pos = joint.position
rc, x_new, y_new = self.convert_joint_coordinates(pos.x, pos.y, pos.z)
return self.joint_s.pack(joint.type,
joint.positionConfidence,
joint.position.x,
joint.position.y,
joint.position.z,
x_new,
y_new)
else:
return self.joint_s.pack(0, 0, 0, 0, 0, 0, 0)
def serialize_skeleton(self, user_frame, frame_index):
if user_frame is not None:
buf = self.frame_s.pack(frame_index, user_frame.timestamp)
users = user_frame.users
if users:
user = users[0]
user_coordinates = user.centerOfMass
state = int(user.skeleton.state)
buf += self.user_s.pack(user.id,
user_coordinates.x,
user_coordinates.y,
user_coordinates.z,
state)
for i in range(15):
buf += self.serialize_joint(user.skeleton[JointType(i)])
else:
buf += self.user_s.pack(0, 0, 0, 0, 0)
for i in range(15):
buf += self.serialize_joint(None)
else:
buf = self.frame_s.pack(0, 0)
buf += self.user_s.pack(0, 0, 0, 0, 0)
for i in range(15):
buf += self.serialize_joint(None)
return buf
def unserialize_hands(self, f):
v = self.frame_s.unpack(f.read(self.frame_s.size))
data = []
for i in range(2):
hands = self.hand_s.unpack(f.read(self.hand_s.size))
data.append(HandDataMock(hands[0], hands[1], hands[2], hands[3], hands[4], hands[5]))
return HandFrameMock(v[0], v[1], data)
def unserialize_joint(self, f):
v = self.joint_s.unpack(f.read(self.joint_s.size))
return JointMock(*v)
def unserialize_skeleton(self, f):
v = []
v += self.frame_s.unpack(f.read(self.frame_s.size))
v += self.user_s.unpack(f.read(self.user_s.size))
joints = []
for i in range(15):
joints.append(self.unserialize_joint(f))
return SkeletonFrameMock(v[0], v[1], v[2], v[3], v[4], v[5], v[6], joints)
class KinectDataReader(object):
def __init__(self, file_name):
super(KinectDataReader, self).__init__()
self.file_name = file_name
self.in_algs_file = open(self.file_name + '.algs', 'rb')
self._s = Serialization()
def readNextFrame(self):
return self._s.unserialize_frame(self.in_algs_file)
Przykładowy skrypt do wczytywania wyników algorytmów śledzenia pozycji anatomicznych (wraz z metadanymi rejestracji):
#!/usr/bin/env python3
# -*- coding: utf-8 -*-
from KinectUtils import KinectDataReader
FILE_PATH = '......'
file_name = '......'
kinect = KinectDataReader(FILE_PATH+file_name)
first_time = 0
joints = []
Time = []
while True:
frame = kinect.readNextFrame()
if frame is None:
print('END OF FILE')
break
frame_index = frame[0]
hands_included = frame[1]
skeleton_included = frame[2]
frame_time = frame[3]
if not first_time:
first_time = frame_time
if skeleton_included:
skel = frame[8]
Time.append(frame_time-first_time)
joints.append(skel.joints)
print('Idx:', frame_index, 'Time:', frame_time-first_time)
print('Head x,y,z [mm]: ', joints[-1][0].x, joints[-1][0].y, joints[-1][0].z)
print('Torso x,y,z [mm]: ', joints[-1][8].x, joints[-1][8].y, joints[-1][8].z)
Plik z metadanymi rejestracji zawiera: nagłówek, wyniki algorytmów śledzenia 15 pozycji anatomicznych, wyniki algorytmów śledzenia pozycji rąk (oddzielna opcja w scenariuszu). Pozycje anatomiczne sylwetki zapisane są w następującej kolejności:
joints = [JOINT_HEAD,
JOINT_NECK,
JOINT_RIGHT_SHOULDER,
JOINT_LEFT_SHOULDER,
JOINT_RIGHT_ELBOW,
JOINT_LEFT_ELBOW,
JOINT_RIGHT_HAND,
JOINT_LEFT_HAND,
JOINT_TORSO,
JOINT_RIGHT_HIP,
JOINT_LEFT_HIP,
JOINT_RIGHT_KNEE,
JOINT_LEFT_KNEE,
JOINT_RIGHT_FOOT,
JOINT_LEFT_FOOT]
Ich położenie (x,y,z) wyrażone jest w jednostkach [mm].
Literatura
- Stone E.E., Butler M., McRuer A., Gray A., Marks J., Skubic M., Evaluation of the Microsoft Kinect for Screening ACL Injury, Conference proceedings: ... Annual International Conference of the IEEE Engineering in Medicine and Biology Society, 2013:4152-5, 2013.
- Webster D., Celik O., Systematic review of Kinect applications in elderly care and stroke rehabilitation, Journal of NeuroEngineering and Rehabilitation, 11:108, 2014.