TI/Programowanie dla Fizyków Medycznych/Obsługa plików graficznych i DICOM: Różnice pomiędzy wersjami
| (Nie pokazano 1 pośredniej wersji utworzonej przez tego samego użytkownika) | |||
| Linia 16: | Linia 16: | ||
==Obsługa plików DICOM - pyDICOM== | ==Obsługa plików DICOM - pyDICOM== | ||
| − | Format plików DICOM, czyli Digital Imaging and Communications in Medicine (Obrazowanie Cyfrowe i Wymiana Obrazów w Medycynie) to norma opracowana przez ACR/NEMA (American College of Radiology / National Electrical Manufacturers Association). Do | + | Format plików DICOM, czyli Digital Imaging and Communications in Medicine (Obrazowanie Cyfrowe i Wymiana Obrazów w Medycynie) to norma opracowana przez ACR/NEMA (American College of Radiology / National Electrical Manufacturers Association). Do obsługi plików tego typu służy między innymi biblioteka pyDICOM. Za jej pomocą wczytujemy przykładowy plik dostępny tutaj [[Media:I00001.dcm]] następującym poleceniem |
<source lang="python"> | <source lang="python"> | ||
import dicom | import dicom | ||
| Linia 25: | Linia 25: | ||
<source lang="python"> | <source lang="python"> | ||
print plik.dir() | print plik.dir() | ||
| − | >>>['AccessionNumber', 'AcquisitionDate', 'AcquisitionDeviceProcessingCode', 'AcquisitionDeviceProcessingDescription', 'AcquisitionNumber', 'AcquisitionTime', 'AnnotationDisplayFormatID', 'BitsAllocated', 'BitsStored', 'BodyPartExamined', 'BorderDensity', 'Columns', 'ContentDate', 'ContentTime', 'ContrastBolusAgent', 'DerivationDescription', 'FilmOrientation', 'HighBit', 'ImageDisplayFormat', 'ImageType', 'ImagerPixelSpacing', 'InstanceNumber', 'InstitutionName', 'Laterality', 'LossyImageCompression', 'Manufacturer', 'Modality', 'NumberOfPoints', 'PatientBirthDate', 'PatientID', 'PatientName', 'PatientOrientation', 'PatientSex', 'PerformedProcedureStepDescription', 'PerformedProcedureStepID', 'PhotometricInterpretation', 'PixelData', 'PixelRepresentation', 'PixelSpacing', 'PlateID', 'PositionerType', 'RefdStudySequence', 'ReferencedStudySequence', 'ReferringPhysicianName', 'RequestAttributesSequence', 'RequestingService', 'RescaleIntercept', 'RescaleSlope', 'RescaleType', 'Rows', 'SOPClassUID', 'SOPInstanceUID', 'SamplesPerPixel', 'Sensitivity', 'SeriesDate', 'SeriesInstanceUID', 'SeriesNumber', 'SeriesTime', 'SpecificCharacterSet', 'StationName', 'StudyDate', 'StudyDescription', 'StudyID', 'StudyInstanceUID', 'StudyTime', 'Trim', 'ViewPosition'] | + | >>> ['AccessionNumber', 'AcquisitionDate', 'AcquisitionDeviceProcessingCode', 'AcquisitionDeviceProcessingDescription', 'AcquisitionNumber', 'AcquisitionTime', 'AnnotationDisplayFormatID', 'BitsAllocated', 'BitsStored', 'BodyPartExamined', 'BorderDensity', 'Columns', 'ContentDate', 'ContentTime', 'ContrastBolusAgent', 'DerivationDescription', 'FilmOrientation', 'HighBit', 'ImageDisplayFormat', 'ImageType', 'ImagerPixelSpacing', 'InstanceNumber', 'InstitutionName', 'Laterality', 'LossyImageCompression', 'Manufacturer', 'Modality', 'NumberOfPoints', 'PatientBirthDate', 'PatientID', 'PatientName', 'PatientOrientation', 'PatientSex', 'PerformedProcedureStepDescription', 'PerformedProcedureStepID', 'PhotometricInterpretation', 'PixelData', 'PixelRepresentation', 'PixelSpacing', 'PlateID', 'PositionerType', 'RefdStudySequence', 'ReferencedStudySequence', 'ReferringPhysicianName', 'RequestAttributesSequence', 'RequestingService', 'RescaleIntercept', 'RescaleSlope', 'RescaleType', 'Rows', 'SOPClassUID', 'SOPInstanceUID', 'SamplesPerPixel', 'Sensitivity', 'SeriesDate', 'SeriesInstanceUID', 'SeriesNumber', 'SeriesTime', 'SpecificCharacterSet', 'StationName', 'StudyDate', 'StudyDescription', 'StudyID', 'StudyInstanceUID', 'StudyTime', 'Trim', 'ViewPosition'] |
</source> | </source> | ||
| − | Podana | + | Podana lista zawiera pola, które posiada wczytany obiekt. W ten sposób mamy dostęp na przykład do nazwiska pacjenta czy daty badania. |
<source lang="python"> | <source lang="python"> | ||
print plik.PatientsName | print plik.PatientsName | ||
| Linia 45: | Linia 45: | ||
print pixel.shape | print pixel.shape | ||
print min(pixel.flatten()),max(pixel.flatten()) | print min(pixel.flatten()),max(pixel.flatten()) | ||
| − | + | py.imshow(pixel,cmap=py.cm.gray,interpolation='nearest') | |
| − | + | py.show() | |
| − | + | >>>(2964, 2364) | |
| − | + | >>> 0 3908 | |
</source> | </source> | ||
[[Plik:dicom1.png]] | [[Plik:dicom1.png]] | ||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
[["Programowanie dla Fizyków Medycznych"]] | [["Programowanie dla Fizyków Medycznych"]] | ||
Aktualna wersja na dzień 12:16, 9 cze 2015
Wczytywanie plików graficznych
Pliki graficzne w większości najpopularniejszych formatów można otworzyć w pythonie korzystając z PIL (Python Image Library). Można to zrobić następującym poleceniem.
from PIL import Image
import numpy as np
import pylab as py
obraz=Image.open('logo2.bmp')
obraz.show()
Jak widać wczytany obiekt nie jest tablicą numpy, ma za to na przykład własną metodę pozwalającą na wyświetlanie wczytanego pliku. Dla naszej wygody możemy jednak przetworzyć tak wczytany plik do postaci tablicy.
im = np.asarray(obraz)
py.imshow(im,cmap = py.cm.gray, interpolation = 'nearest')
py.show()
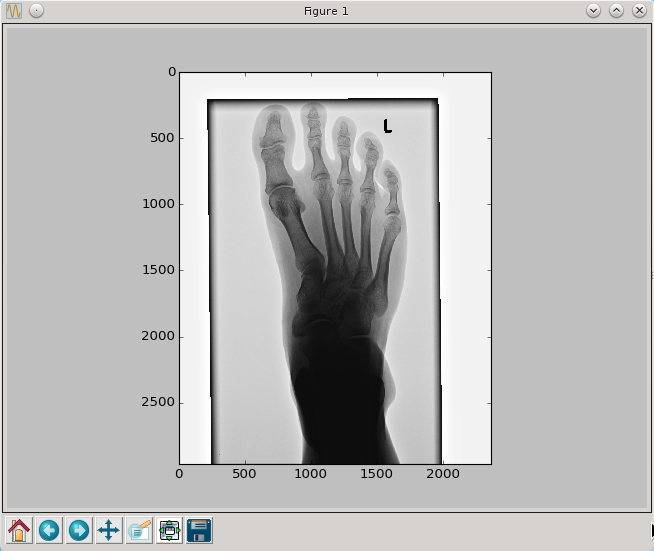
Obsługa plików DICOM - pyDICOM
Format plików DICOM, czyli Digital Imaging and Communications in Medicine (Obrazowanie Cyfrowe i Wymiana Obrazów w Medycynie) to norma opracowana przez ACR/NEMA (American College of Radiology / National Electrical Manufacturers Association). Do obsługi plików tego typu służy między innymi biblioteka pyDICOM. Za jej pomocą wczytujemy przykładowy plik dostępny tutaj Media:I00001.dcm następującym poleceniem
import dicom
plik=dicom.read_file('I00001.dcm')
Plik DICOM poza danymi samego obrazu medycznego zawiera wiele dodatkowych informacji na temat przeprowadzonego badania. Listę dostępnych danych możemy uzyskać poleceniem
print plik.dir()
>>> ['AccessionNumber', 'AcquisitionDate', 'AcquisitionDeviceProcessingCode', 'AcquisitionDeviceProcessingDescription', 'AcquisitionNumber', 'AcquisitionTime', 'AnnotationDisplayFormatID', 'BitsAllocated', 'BitsStored', 'BodyPartExamined', 'BorderDensity', 'Columns', 'ContentDate', 'ContentTime', 'ContrastBolusAgent', 'DerivationDescription', 'FilmOrientation', 'HighBit', 'ImageDisplayFormat', 'ImageType', 'ImagerPixelSpacing', 'InstanceNumber', 'InstitutionName', 'Laterality', 'LossyImageCompression', 'Manufacturer', 'Modality', 'NumberOfPoints', 'PatientBirthDate', 'PatientID', 'PatientName', 'PatientOrientation', 'PatientSex', 'PerformedProcedureStepDescription', 'PerformedProcedureStepID', 'PhotometricInterpretation', 'PixelData', 'PixelRepresentation', 'PixelSpacing', 'PlateID', 'PositionerType', 'RefdStudySequence', 'ReferencedStudySequence', 'ReferringPhysicianName', 'RequestAttributesSequence', 'RequestingService', 'RescaleIntercept', 'RescaleSlope', 'RescaleType', 'Rows', 'SOPClassUID', 'SOPInstanceUID', 'SamplesPerPixel', 'Sensitivity', 'SeriesDate', 'SeriesInstanceUID', 'SeriesNumber', 'SeriesTime', 'SpecificCharacterSet', 'StationName', 'StudyDate', 'StudyDescription', 'StudyID', 'StudyInstanceUID', 'StudyTime', 'Trim', 'ViewPosition']
Podana lista zawiera pola, które posiada wczytany obiekt. W ten sposób mamy dostęp na przykład do nazwiska pacjenta czy daty badania.
print plik.PatientsName
print plik.AcquisitionDate
>>>GUBIEC^TOMASZ
>>>20141119
Dane te możemy dodatkowo edytować.
plik.PatientsName='Jan Kowalaski'
print plik.PatientsName
>>>Jan Kowalaski
Najciekawsze jednak dla nas będą dane opisujące sam obraz. Możemy uzyskać do nich dostęp w wygodnej dla nas postaci tablicy numpy.
pixel=plik.pixel_array
print pixel.shape
print min(pixel.flatten()),max(pixel.flatten())
py.imshow(pixel,cmap=py.cm.gray,interpolation='nearest')
py.show()
>>>(2964, 2364)
>>> 0 3908