Uczenie maszynowe i sztuczne sieci neuronowe/Ćwiczenia 11: Różnice pomiędzy wersjami
(Utworzono nową stronę "=Algorytm k-means= Algorytm k-means jest zaimplementowany w module <tt>scipy.cluster.vq</tt> (vq: vector quantization) (http://docs.scipy.org/doc/scipy/reference/clust...") |
m |
||
| Linia 1: | Linia 1: | ||
| + | [[Uczenie_maszynowe_i_sztuczne_sieci_neuronowe_cw]]/Uczenie bez nadzoru | ||
| + | |||
=Algorytm k-means= | =Algorytm k-means= | ||
Algorytm k-means jest zaimplementowany w module <tt>scipy.cluster.vq</tt> (vq: vector quantization) ([[http://docs.scipy.org/doc/scipy/reference/cluster.vq.html dokumentacja]]). Mamy tam funkcję | Algorytm k-means jest zaimplementowany w module <tt>scipy.cluster.vq</tt> (vq: vector quantization) ([[http://docs.scipy.org/doc/scipy/reference/cluster.vq.html dokumentacja]]). Mamy tam funkcję | ||
Aktualna wersja na dzień 17:19, 21 mar 2018
Uczenie_maszynowe_i_sztuczne_sieci_neuronowe_cw/Uczenie bez nadzoru
Spis treści
Algorytm k-means
Algorytm k-means jest zaimplementowany w module scipy.cluster.vq (vq: vector quantization) ([dokumentacja]). Mamy tam funkcję
kmeans(obs, k_or_guess, iter=20, thresh=1e-05)
optymalizującą położenia centroidów, oraz pomocniczą funkcję
vq
przypisującą poszczególne obserwacje do skupisk reprezentowanych przez centroidy.
Przed zapuszczeniem algorytmu k-means na danych dobrze jest przeskalować każdą z cech w macierzy wejściowej, tak aby miała jednostkową wariancję. Można to zrobić za pomoca funkcji whiten.
Przykładowy kod.
Kod ten pokazuje jak:
- wygenerować symulowane dane
- przeskalować je, tak aby miały jednostkową wariancję w każdej z cech.
- podzielić je na dwa skupiska
- zilustrować wynik
from pylab import plot,show
from numpy import vstack,array
from numpy.random import rand
from scipy.cluster.vq import kmeans,vq,whiten
# generujemy dane:
# - 150 dwuwymiarowych punktów z rozkładu jednorodnego ze średnią (1,1)
# - 150 dwuwymiarowych punktów z rozkładu jednorodnego ze średnią (0.5,0.5)
data = vstack((rand(150,2) + array([.5,.5]),rand(150,2)))
data = whiten(data)
# policz K-Means dla K = 2 (2 skupiska)
centroids,_ = kmeans(data,2)
# przypisz wektory wejściowe do skupisk
idx,_ = vq(data,centroids)
# narysuj wyniki
plot(data[idx==0,0],data[idx==0,1],'ob',
data[idx==1,0],data[idx==1,1],'or')
plot(centroids[:,0],centroids[:,1],'sg',markersize=8)
show()
Segmentacja obrazu algorytmem k-means
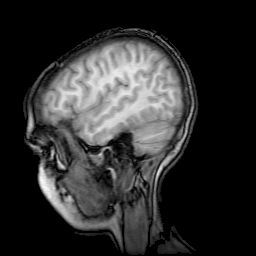
W tym ćwiczeniu zapoznamy się z zastosowaniem algorytmu analizy skupień do segmetacji obrazu. Segmentacja tegotypu może stanowic etap wstępnego przetwarzania na potrzeby np. detekcji obiektów lub klasyfikacji. W zadaniu tym zapoznamy sie także z metodą dobierania atumatycznie ilości skupisk.
Obrazek na którym będziemy pracować, proszę go zapisać w bieżącym katalogu:
Najpierw importujemy i oglądamy obrazek:
from pylab import plot,show,figure,imshow,cm, imread, axis
import numpy as np
from scipy.cluster.vq import kmeans,vq
im = imread('Skan.png')
# Oryginalny obrazek miał przestrzeń barwną RGB.
# Spłaszczamy przestrzeń barwną obrazka
im = im.mean(axis=2)
#oglądamy obrazek
imshow(im, cmap=cm.gray)
axis('off')
show()
imshow(im, cmap=cm.gray)
axis('off')
show()
Teraz zamieniamy rysunek (dwuwymiarowa tablica 256x256) na wektor (o długości 256*256 )
data = im[:]
data.shape = 256*256,1
Teraz będziemy próbować podzielić ten wektor na skupiska (w liczbie od 2 do 9). Dla każdej konkretnej ilości skupisk obliczamy dwie wielkości:
- [math]J_{intra}(K)[/math] - to jest miara odległości wewwnątrz centrów: równanie na J
- [math]J_{inter}(K) = \min_{j\lt i} \sqrt{ (\mu_i -\mu_j)^T(\mu_i - \mu_j)} [/math] : to najmniejsza odległości między centrami
K_max = 9
J_inter = np.ones(K_max)*1e16
J_intra = np.zeros(K_max)
centroids =[]
for K in range(2,K_max):
trial =0
while (len(centroids)<K)&(trial<20):
centroids,J_intra[K] = kmeans(data,K)
trial+=1
print 'K: ',K, len(centroids)
for ki in range(len(centroids)):
for kj in range(ki):
print ki, kj
print centroids[ki]
print centroids[kj]
################
## dopisz kod obliczający odległość między centrami i oznacz ją d
################
# jeśli uzyskana odległość jest mniejsza niż dotychczas zapamiętana to ją zapamiętujemy:
if J_inter[K]>d:
J_inter[K]=d
print K, J_intra[K],J_inter[K]
Wykreślamy stosunek [math],J_{intra}/J_{inter}[/math] i znajdujemy K dla którego jest najmniejszy:
figure(1)
plot(range(2,K_max),J_intra[2:]/J_inter[2:])
K_opt = np.argmin(J_intra[2:]/J_inter[2:])+2
print K_opt
Dla tej optymalnej ilości skupisk znajdujemy położenia centrów i przypisujemy klasę dla każdego punktu danych:
centroids,J_intra[K] = kmeans(data,K_opt)
# przypisujemy klasę
idx,_ = vq(data,centroids)
Formatujemy wektor w obrazek i podziwiamy efekt:
idx.shape = 256,256
figure(2)
imshow(idx, cmap=cm.gray)
show()
Dla porównania proszę wykreślić histogram odcieni szarości dla wekotra data.
Algorytm EM: implementacja
W celu zapoznania się z algorytmem EM zaimplementujemy go.
Program, który powstanie po uzupełnieniu kodu powinien ilustrować dopasowywanie modelu EM do danych będzących sumą dwóch rozkładów gaussowskich:
Funkcje pomocnicze
Najpierw standardowe importy i kilka funkcji pomocniczych:
# -*- coding: utf-8 -*-
import matplotlib
matplotlib.use('TkAgg')
import pylab as py
import random, copy
import numpy as np
import sys
def pnorm(x, m, s):
"""
Oblicza gęstość wielowymiarowego rozkładu normalnego dla punktów
w wektorze x
Parametry rozkładu :
m - średnia
s- macierz kowariancji
dla zwiększenia czytelności kodu stosujemy typ matrix
"""
xm = np.matrix(x-m)
xmt = np.matrix(x-m).transpose()
for i in xrange(len(s)): # zabezpieczenie dla większej stabliności numerycznej
if s[i,i] <= sys.float_info[3]: # min float
s[i,i] = sys.float_info[3]
sinv = np.linalg.inv(s)
return (2.0*np.pi)**(-len(x)/2.0)*(1.0/np.sqrt(np.linalg.det(s)))\
*np.exp(-0.5*(xm*sinv*xmt))
def draw_params(t,nbclusters):
'''funkcja do losowania parametrów początkowych
t - zbiór treningowy
'''
nbobs,nbfeatures = t.shape
# inicjuje średnie przez losowanie punktu ze zbioru danych
tmpmu = np.array([t[random.uniform(0,nbobs),:]],np.float64)
# kowariancje inicjowane są jako macierze diagonalne , wariancja dla każdej cechy inicjowana jest jako wariancja tej cechy dla całego zbioru
sigma = np.zeros((nbfeatures,nbfeatures))
for f in range(nbfeatures):
sigma[f,f] = np.var(t[:,f])
#phi inicjujemy tak, że każda składowa mieszanki ma takie samee prawdopodobieństwo
phi = 1.0/nbclusters
print 'INIT:',tmpmu,sigma,phi
return {'mu': tmpmu,\
'sigma': sigma,\
'phi': phi}
def plot_gauss(mu,sigma):
''' Funkcja rysująca kontury funkcji gęstości prawdopodobieństwa
dwuwymiarowego rozkładu Gaussa'''
x = np.arange(-6.0, 6.0001, 0.1)
y = np.arange(-6.0, 6.0001, 0.1)
X,Y = np.meshgrid(x, y)
X.shape = 1,len(x)*len(y)
Y.shape = 1,len(x)*len(y)
P = np.vstack((X,Y))
invS = np.linalg.inv(sigma)
R = P.T-mu
z = np.zeros(len(R))
for i in range(len(R)):
z[i] = np.exp(-0.5*np.dot( R[i,:].T,np.dot(invS,R[i,:])))
z.shape = len(x),len(y)
py.contourf(x,y,z,alpha = 0.5)
py.plot(mu[0],mu[1],'o')
Szkielet algorytmu
Poniższy kod to szkielet właściwej funkcji wykonującej optymalizację. Trzeba go uzupełnić implementując równania z wykładu. Proszę uważnie czytać komentarze.
def expectation_maximization(t, nbclusters=2, nbiter=3, normalize=False,\
epsilon=0.001, monotony=False, datasetinit=True):
"""
t - zbiór treningowy,
Każdy wiersz t jest przykładem (obserwacją), każda kolumna to cecha
'nbclusters' ilość klastrów, z których budujemy model mieszany
'nbiter' ilość iteracji
'epsilon' kryterium zbieżności
Powtórz kroki E i M aż do spełnienia warunku |E_t - E_{t-1}| < ε
Funkcja zwraca parametry modelu (centra i macerze kowariancji Gaussów i ich wagi \phi) oraz
etykiety punktów zbioru treningowego oznaczające do którego z Gaussów w modelowanej mieszance należą.
"""
nbobs,nbfeatures = t.shape
### Opcjonalna normalizacja
if normalize:
for f in xrange(nbfeatures):
t[:,f] /= np.std(t[:,f])
result = {}
random.seed()
# szykujemy tablice na prawdopodobieństwa warunowe
Pz = np.zeros((nbobs,nbclusters)) # P(z|x): opisywane równaniami (2) i (3) z wykładu
Px = np.zeros((nbobs,nbclusters)) # P(x|z): opisywane równaniem (4)
# inicjujemy parametry dla każdego składnika mieszankni
# params będzie listą taką, że params[i] to słownik
# zawierający parametry i-tego składnika mieszanki
params = []
for i in xrange(nbclusters):
params.append( draw_params(t,nbclusters) )
old_log_estimate = sys.maxint # init
log_estimate = sys.maxint/2 + epsilon # init
estimation_round = 0
# powtarzaj aż zbiegniesz
while (abs(log_estimate - old_log_estimate) > epsilon\
and (not monotony or log_estimate < old_log_estimate)):
restart = False
old_log_estimate = log_estimate
########################################################
# krok E: oblicz Pz dla każdego przykładu (czyli w oznaczeniach z wykładu w_i^j)
########################################################
# obliczamy prawdopodobieństwa Px[j,i] = P(x_j|z_j=i)
for j in xrange(nbobs): # iterujemy po przykładach
for i in xrange(nbclusters): # iterujemy po składnikach
Px[j,i] = pnorm(t[j,:], params[i]['mu'], params[i]['sigma']) # (równanie 4)
# obliczamy prawdopodobieństwa Pz[j,i] = P(z_j=i|x_j)
# najpierw licznik równania (3)
for j in xrange(nbobs):
for i in xrange(nbclusters):
Pz[j,i] = .............*params[i]['phi']
# mianownik równania (3)
for j in xrange(nbobs):
tmpSum = 0.0
for i in xrange(nbclusters):
tmpSum += ..................
# składamy w całość Pz[j,i] = P(z_j=i|x_j)
Pz[j,:] /= tmpSum
###########################################################
# krok M: uaktualnij paramertry (sets {mu, sigma, phi}) #
###########################################################
#print "iter:", iteration, " estimation#:", estimation_round,\
# " params:", params
for i in xrange(nbclusters):
print "------------------"
# parametr phi: równanie (6)
Sum_w = np.sum(Pz[:,i])
params[i]['phi'] = Sum_w/nbobs
if params[i]['phi'] <= 1.0/nbobs: # restartujemy jeśli zanika nam któraś składowa mieszanki
restart = True
print "Restarting, p:",params[i]['phi']
break
print 'i: ',i,' phi: ', params[i]['phi']
# średnia: równanie (7)
m = np.zeros(nbfeatures)
for j in xrange(nbobs):
m += ......................
params[i]['mu'] = m/Sum_w
print 'i: ',i,' mu: ', params[i]['mu']
# macierz kowariancji: równanie (8)
s = np.matrix(np.zeros((nbfeatures,nbfeatures)))
for j in xrange(nbobs):
roznica = np.matrix(t[j,:]-params[i]['mu'])
s += Pz[j,i]*(roznica.T*roznica)
params[i]['sigma'] = s/Sum_w
print params[i]['sigma']
### Testujemy czy składniki się nie sklejają i w razie potrzeby restartujemy
if not restart:
restart = True
for i in xrange(1,nbclusters):
if not np.allclose(params[i]['mu'], params[i-1]['mu'])\
or not np.allclose(params[i]['sigma'], params[i-1]['sigma']):
restart = False
break
if restart:
old_log_estimate = sys.maxint # init
log_estimate = sys.maxint/2 + epsilon # init
params = [draw_params(t,nbclusters) for i in xrange(nbclusters)] # losujemy nowe parametry startowe
print 'RESTART'
continue
####################################
# liczymy estymatę log wiarygodności: równaie (1) #
####################################
log_estimate = np.sum([np.log(np.sum(\
[Px[j,i]*params[i]['phi'] for i in xrange(nbclusters)]))\
for j in xrange(nbobs)])
print "(EM) poprzednia i aktualna estymata log wiarygodności: ",\
old_log_estimate, log_estimate
estimation_round += 1
##########################
# rysujemy aktualny stan modelu
##########################
py.ioff()
py.clf()
py.ion()
for i in xrange(nbclusters):
plot_gauss(np.array(params[i]['mu']),np.array(params[i]['sigma']))
py.plot(x[:,0],x[:,1],'g.')
py.axis('equal')
py.draw()
# Pakujemy wyniki
result['quality'] = -log_estimate
result['params'] = copy.deepcopy(params)
result['clusters'] = [[o for o in xrange(nbobs)\
if Px[o,c] == max(Px[o,:])]\
for c in xrange(nbclusters)]
return result
Program
Przykładowy program korzystający z powyższych funkcji:
##########################################
# PROGRAM
#########################################
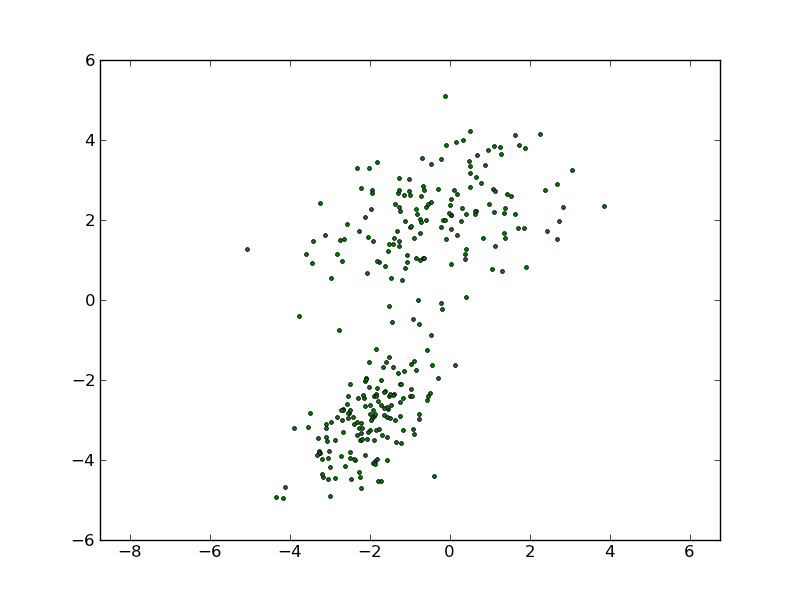
# robimy mieszankę dwóch gaussów:
#parametry rozkładu
# wektor średnich:
mu1 = [-2,-3]
# macierz kowariancji:
Sigma1 = np.array([[1, 0.5],
[0.5, 1]])
# generujemy dane:
x1 = np.random.multivariate_normal(mu1, Sigma1, 150) #
mu2 = [-0.5,2]
# macierz kowariancji:
Sigma2 = np.array([[3, 0.5],
[0.5, 1]])
# generujemy dane:
x2 = np.random.multivariate_normal(mu2, Sigma2, 150) #
# łączymy x1 i x2 aby otrzymac jeden zbiór
x = np.vstack((x1,x2))
py.plot(x[:,0],x[:,1],'g.')
py.axis('equal')
py.show()
py.figure()
res = expectation_maximization(x, nbclusters=2, nbiter=3, normalize=False,\
epsilon=0.001, monotony=False, datasetinit=True)
py.ioff()
py.show()
# wypisz parametry
print 'Dopasowany model: '
print res['params']
Aby obliczyć gęstość prawdopodobieństwa rozkładu mieszanego dla pewnego nowego punktu "x" możemy zastosować poniższą funkcję:
def prob_mix(params, x):
'''params - parametry dopasowanego gaussowskiego modelu mieszanego
x - punkt wejścowy,
funkcja zwraca gestość prawdopodobieństwa, dla x w rozkładzie mieszanym
'''
prob = 0
for i in range(len(params)):
prob+= pnorm(x, params[i]['mu'], params[i]['sigma']) * params[i]['phi']
return prob
#---------------- przykładowe użycie: ----------------
x=(6,-4)
print 'P(x=(',str(x),')):', prob_mix(res['params'], x)