WnioskowanieStatystyczne/Elementy statystyki wielowymiarowej: Różnice pomiędzy wersjami
| Linia 87: | Linia 87: | ||
f(t)=ke^{-\frac{1}{2}(t-\mu )A(t-\mu )^{T}} | f(t)=ke^{-\frac{1}{2}(t-\mu )A(t-\mu )^{T}} | ||
</math> | </math> | ||
| + | |||
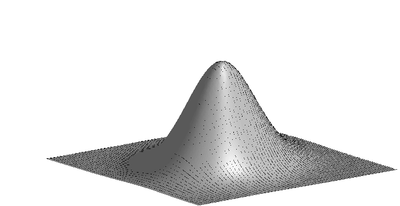
| + | [[Plik:gauss2d.png|thumb|400px|center|Dwumianowy rozkład normalny, wartość prawdopodobieństwa jako wysokość nad płaszczyzną]] | ||
<math>t=(x,y)-</math> wektor zmiennej losowej | <math>t=(x,y)-</math> wektor zmiennej losowej | ||
| Linia 110: | Linia 112: | ||
\right] | \right] | ||
</math> | </math> | ||
| − | |||
| − | |||
==Analiza wariancji wielu zmiennych (''Multivariate ANalysis of VAriance — MANOVA'')== | ==Analiza wariancji wielu zmiennych (''Multivariate ANalysis of VAriance — MANOVA'')== | ||
Wersja z 10:54, 29 maj 2017
Wnioskowanie_Statystyczne_-_wykład
Spis treści
- 1 Elementy statystyki wielowymiarowej
- 1.1 Kowariancja i współczynnik korelacji
- 1.2 Macierz kowariancji
- 1.3 Dwumianowy rozkład normalny
- 1.4 Analiza wariancji wielu zmiennych (Multivariate ANalysis of VAriance — MANOVA)
- 1.5 Analiza składowych głównych (Principal Components Analysis, PCA)
- 1.6 Analiza dyskryminacyjna (Discriminant Analysis )
- 1.7 Analiza czynnikowa (Factor Analysis )
- 1.8 Analiza skupień — Cluster Analysis
Elementy statystyki wielowymiarowej
Przypomnijmy najpierw pojęcia
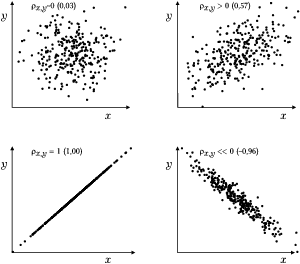
Kowariancja i współczynnik korelacji
Miarą związku między zmiennymi [math]x[/math] i [math]y[/math] jest kowariancja
[math] \sigma_{x, y} = E\left( (x-\mu_{x})(y-\mu_{y})\right) [/math]
lub unormowany do jedności współczynnik korelacji zmiennych [math]x[/math] i [math]y[/math]:
[math] \rho_{x, y}= \frac{\sigma_{x, y}}{\sigma_x \sigma_y}= \frac{E\left( \left(x-\mu_{x})(y-\mu_{y}\right)\right)} {\sqrt{E\left( (x-\mu_{x})^2\right) E\left( (y-\mu_{y})^2\right)}}, [/math]
gdzie [math]\mu_x[/math] i [math]\mu_y[/math] to odpowiednio wartości oczekiwane zmiennych [math]x[/math] i [math]y[/math]. Jeśli zmienne [math]x[/math] i [math]y[/math] związane są deterministyczną zależnością liniową (typu [math]y=c_1 x+c_2[/math]), to ich korelacja wynosi [math]1[/math] (lub [math]-1[/math], jeśli [math]c_1\lt 0[/math]). Jeśli wzrostowi zmiennej [math]x[/math] towarzyszy statystycznie wzrost zmiennej [math]y[/math], to ich korelacja jest dodatnia (pomiędzy [math]0[/math] a [math]1[/math]). Dla zmiennych niezależnych korelacja wynosi [math]0[/math].
Macierz kowariancji
[math] C=E[(x-\mu )(x-\mu )^{T}], \qquad \\ c_{ij}=E[(x_{i}-\mu_{i})(x_{j}-\mu _{j})] [/math]
dla [math]x=(x_{1,}x_{2})[/math] i [math]\mu =(\mu _{1}, \mu _{2})[/math]
[math] C=E\left[ \left(\begin{matrix}{x_{1}-\mu _{1}}\\ {x_{2}-\mu _{2}} \end{matrix} \right) \left( x_{1}-\mu_{1},x_{2}-\mu _{2}\right) \right] = [/math]
[math] \left[ \begin{matrix} E\left[ \left( x_{1}-\mu _{1}\right) ^{2}\right] & E\left[ \left( x_{1}-\mu _{1}\right) \left( x_{2}-\mu _{2}\right) \right] \\ E\left[ \left( x_{2}-\mu _{2}\right) \left( x_{1}-\mu _{1}\right) \right] & E \left[ \left( x_{2}-\mu _{2}\right) ^{2}\right] \end{matrix} \right] = [/math]
[math]
\left[
\begin{matrix}
\sigma _{1}^{2} & \sigma _{12} \\
\sigma _{21} & \sigma _{2}^{2}
\end{matrix}
\right]
\end{matrix}
[/math]
Interpretacja współczynnika korelacji znajduje się pod hasłem Regresja liniowa.
Dwumianowy rozkład normalny
[math] f(t)=ke^{-\frac{1}{2}(t-\mu )A(t-\mu )^{T}} [/math]
[math]t=(x,y)-[/math] wektor zmiennej losowej
[math]\mu =(\mu _{1},\mu _{2})-[/math] wektor wartości oczekiwanych
[math]k-[/math] stała normalizjąca
[math]A-[/math] odwrotność macierzy kowariancji [math]C[/math]
[math] A=\left[ \begin{matrix} \sigma _{x}^{2} & \sigma _{xy} \\ \sigma _{xy} & \sigma _{y}^{2} \end{matrix} \right] ^{-1}=\frac{1}{\sigma _{x}^{2}\sigma _{y}^{2}-\left( \sigma _{xy}\right) ^{2}}\left[ \begin{matrix} \sigma _{y}^{2} & -\sigma _{xy} \\ -\sigma _{xy} & \sigma _{x}^{2} \end{matrix} \right] [/math]
Analiza wariancji wielu zmiennych (Multivariate ANalysis of VAriance — MANOVA)
Zmienna losowa [math]X[/math] opisywana wektorem ([math]x_{1},...,x_{k})[/math], podobnie wartość średnia staje się wektorem o tym samym wymiarze: ([math] \mu _{1},...,\mu _{k}).[/math] Macierz kowariancji (%i ???) zmiennej losowej
[math] S=\left[ \begin{matrix} \upsilon ar(x_{1}) & co\upsilon (x_{1},x_{2}) & ... & co\upsilon (x_{1},x_{k}) \\ co\upsilon (x_{2},x_{1}) & \upsilon ar(x_{2}) & ... & co\upsilon (x_{2},x_{k}) \\ ... & ... & ... & ... \\ co\upsilon \left( x_{k},x_{1}\right) & co\upsilon (x_{k},x_{2}) & ... & \upsilon ar(x_{k}) \end{matrix} \right] [/math]
gdzie:
[math] \upsilon ar(x_{i})=\sigma _{x_{i}}=E((x_{i}-\mu _{i})^{2}) [/math]
[math] co\upsilon (x_{i},x_{k})=\sigma _{x_{i},x_{k}}=E((x_{i}-\mu _{i})(x_{k}-\mu _{k})) [/math]
Wielowymiarowy rozkład normalny
[math] \Phi (X)=\frac{1}{\sqrt{(2\pi )^{k}}\sqrt{\left| S\right| }}e^{-\frac{(X-\mu )^{\prime }S^{-1}(X-\mu )}{2}} [/math]
Od tego momentu przyjmujemy, że zmienne [math]X[/math] podlegają takiemu właśnie rozkładowi. Jeśli [math]X[/math] pochodzą z próby podzielonej na grupy, to podobnie jak w [math]ANOVA[/math] możemy skonstruować macierze wariancji wewnątrzgrupowych i międzygrupowych i dowieść, że [math]S=S_{wew}+S_{pom}[/math]. Iloraz wyznaczników macierzy [math]S_{wew}[/math] i [math]S[/math] podlega rozkładowi [math]\Lambda [/math] Wilksa:
[math] \Lambda =\frac{\left| S_{wew}\right| }{\left| S\right| }=\frac{\left| S_{wew}\right| }{\left| S_{wew}+S_{pom}\right| } [/math]
i służy testowaniu hipotezy o braku różnic między grupami. Ogólnie statystyki Wilksa można używać do testowania hipotezy [math]h[/math] w postaci
[math] \Lambda =\frac{\left| S_{wew}\right| }{\left| S_{wew}+S_{h}\right| } [/math]
gdzie [math]S_{h}[/math] — macierz kowariancji odpowiadająca testowanej hipotezie.
Analiza składowych głównych (Principal Components Analysis, PCA)
Przedstawiamy macierz kowariancji w postaci diagonalnej
[math] S=\left[ \begin{matrix} r_{11} & r_{12} & ... & r_{1k} \\ r_{21} & r_{22} & ... & r_{2k} \\ ... & ... & ... & ... \\ r_{k1} & r_{k2} & ... & r_{kk} \end{matrix} \right] \left[ \begin{matrix} \lambda _{1} & 0 & ... & 0 \\ 0 & \lambda _{2} & ... & 0 \\ ... & ... & ... & ... \\ 0 & 0 & ... & \lambda _{k} \end{matrix} \right] \left[ \begin{matrix} r_{11} & r_{21} & ... & r_{k1} \\ r_{12} & r_{22} & ... & r_{k2} \\ ... & ... & ... & ... \\ r_{1k} & r_{2k} & ... & r_{kk} \end{matrix} \right] [/math]
Wielkości [math]\lambda _{i}[/math] są rozwiązaniami równania [math]\left| S-\lambda I\right| =0,[/math] a wektor [math]r_{i}[/math] osiami nowego układu współrzędnych. Składowe [math]PCA[/math] są liniowymi kombinacjami obserwowanych zmiennych.
Analiza dyskryminacyjna (Discriminant Analysis )
Wielowymiarowe wektory próby [math]X[/math] mamy podzielone na grupy, szukamy funkcji najlepiej je rozdzielającej, która umożliwi zaklasyfikowanie nowej obserwacji. Rozdzielenie grup odpowiada w przypadku jednowymiarowym maksymalizacji stosunku wariancji międzygrupowej do wariancji wewnątrzgrupowej
[math] F=\frac{\left( n-k\right) s_{pom}}{\left( k-1\right) s_{wew}} [/math]
W przypadku wielowymiarowym mamy do czynienia z macierzami kowariancji; możemy rozpatrywać wielkość
[math] F_{a}=\frac{a^{\prime }S_{pom}a}{a^{\prime }S_{wew}a} [/math]
Maksymalizacja tej wielkości względem [math]a[/math] daje wektor własny macierzy [math]S_{wew}^{-1}S_{pom}[/math] odpowiadający największej wartości własnej. Wektory własne odpowiadające kolejnym wartościom własnym zwiemy współrzędnymi dyskryminacyjnymi, tworzącymi przestrzeń dyskryminacyjną.
Analiza czynnikowa (Factor Analysis )
opiera się na założeniu istnienia ukrytych czynników, stara się przedstawić obserwowane zmienne w postaci:
obserwowana zmienna = liniowa kombinacja czynników + błąd
w odróżnieniu od PCA, realizującej model
składowa = liniowa kombinacja obserwowanych zmiennych
Analiza skupień — Cluster Analysis
Wejściem dla tej procedury jest zestaw danych, a wyjściem ich podział na grupy. Można go zrealizować na wiele sposobów: [math]N[/math] punktów [math]x^{1}...x^{N},[/math] z których każdy opisany jest przez [math]k[/math] cech [math]x_{1}...x_{k}[/math].
Metody polegające na kolejnym łączeniu punktów
Startujemy z N klastrów jednopunktowych, w każdym kroku łączymy najbliższe. Wynikiem działania jest drzewo łączenia, na którym sami musimy wybrać ilość klastrów. Wynik zależy silnie od przyjętych definicji odległości między klastrami oraz definicji odległości między punktami.
Odległości między punktami:
- Odległość Euklidesowa [math]d(x,y)=\sqrt{\underset{i=1}{\overset{k}{\sum }}(x_{i}-y_{i})^{2}}[/math] (czuła na różne skale cech).
- Odległość korelacyjna [math]d(x,y)=1-\rho (x,y),[/math] gdzie [math]\rho (x,y)=\frac{\underset{i=1}{\overset{k}{\sum }}(x-\overline{x})(y- \overline{y})}{\sigma _{x}\sigma _{y}} [/math] (znormalizowana do przedziału (0,2), mniejsza im lepiej skorelowane punkty).
Odległości między klastrami:
- Najbliższego sąsiada (single linkage) - odległość między dwoma najbliższymi elementami klastrów [math]A[/math] i [math]B[/math]: [math]d(A,B)=\min_{x,y}d(x,y),\ \ \ \ \ x\in A,\ y\in B[/math]
- (complete linkage ) - odległość między dwoma najbliższymi elementami klastrów [math]A[/math] i [math]B[/math]: [math]d(A,B)=\max_{x,y}d(x,y),\ \ \ \ \ x\in A,\ y\in B [/math]
- (centroid) - odległość między środkami klastrów,
- (avarage) - średnia odległości, itd...
Metoda K–średnich (K – means )
Wybieramy ilość klastrów, podział dokonywany jest w iteracyjnej procedurze dążącej do minimalizacji stosunku wariancji pomiędzy klastrami do wariancji wewnątrz klastrów - niejako [math]ANOVA[/math] bez ustalonego wstępnie przyporządkowania, maksimum [math] F[/math] poszukiwane drogą przemieszczania elementów między klastrami.